요약
대형 언어 모델(large language model, LLM)의 활용이 의료 분야에서 빠르게 확대되면서, 표준화된 보고 지침의 필요성이 커지고 있다. 이 논문에서는 LLM을 활용한 연구를 위한 다변수 예측모델의 투명한 보고(TRIPOD-LLM) 지침을 제시하였다. TRIPOD-LLM은 기존 TRIPOD와 인공지능(artificial intelligence) 확장 지침을 기반으로 하며, 바이오 메디컬 분야에서 LLM이 가지는 고유한 도전 과제들을 반영하고 있다. 이 지침은 제목부터 논의까지 주요 내용을 포괄하는 19개 주요 항목과 50개 세부 항목으로 구성되어 있다. 다양한 LLM 연구설계와 작업에 적용할 수 있도록 모듈형 형식을 도입하였고, 모든 연구에 공통적으로 적용할 수 있는 14개 주요 항목과 32개 세부 항목을 포함한다. 이 지침은 신속한 델파이(Delphi) 과정과 전문가 합의를 거쳐 개발하였으며, 투명성과 인간 감독, 과업 특이적 성과(task-specific performance) 보고의 중요성을 강조한다. 또한 지침의 손쉬운 작성과 제출용 PDF 생성을 지원하는 인터랙티브 웹사이트(
https://tripod-llm.vercel.app/)를 소개한다. TRIPOD-LLM은 ‘생명력 있는 문서’로서, 연구현장의 변화에 맞추어 지속적으로 개정될 예정이다. 이 지침을 통해 LLM 연구의 보고 수준을 높이고, 재현성과 임상 적용 가능성을 강화하는 데 기여하려고 한다.
서론
의료 분야에서 대형 언어 모델(large language model, LLM)의 도입은 계속 확대되고 있으며, 현재는 물론 미래에도 행정 및 의료서비스 제공 등 다양한 영역에서의 활용 가능성이 논의되고 있다. 대표적인 예로, 환자 커뮤니케이션 초안 생성, 의료문서 요약, 질의응답, 정보검색, 의료진단, 치료 권고, 환자교육, 의학교육 등이 있다[
1-
5]. LLM의 빠른 발전이 기존 규제 및 거버넌스 체계를 한계까지 밀어붙여, 이러한 복잡한 범용모델을 완전하게 반영하지 못하는 임시방편적인 해결책들이 나타나고 있다[
6-
8]. 더 나아가 LLM 개발이 가속화되면서 학술지 및 전문가 심사(peer review), 출판일정, 시의적절한 지침을 제공해야 하는 규제기관들도 어려움을 겪고 있다. 이에 대응하기 위해 연구자들은 프리프린트(preprint)를 빠르게 발표하고 있고, 보고할 때도 임시방편적 접근을 택하는 경우가 많다.
보고 지침(reporting guideline)은 연구의 표준화와 투명한 보고, 동료평가 절차를 위한 확장 가능한 틀을 제공한다. 중요한 예시로, 진단 및 예후예측모델 연구를 위한 최소 보고 기준을 확립하고자 2015년에 처음 도입된 transparent reporting of a multivariable model for individual prognosis or diagnosis (TRIPOD) 이니셔티브가 있다(
https://www.tripod-statement.org) [
9]. TRIPOD는 건강연구 보고의 질과 투명성 향상을 목표로 하는 국제적 노력인 enhancing the quality and transparency of health research (EQUATOR) 네트워크의 핵심 지침 중 하나이다[
10]. TRIPOD는 여러 학술지가 폭넓게 지지 및 권고하고 있으며, 저자 안내문에도 자주 포함되어 있다. 이후 TRIPOD는 인공지능(artificial intelligence, AI)의 실질적 발전에 대응하여, 머신러닝(machine learning) 분야의 최신 모범 사례를 반영한 TRIPOD+AI로 업데이트되었다[
11]. 또한 모델 수명 주기 전반에 걸친 AI 개발을 위한 보완적 지침들도 제시되어 왔다[
12-
14].
LLM은 AI 내에서도 독특한 특성을 지닌 새로운 영역으로, 기존 TRIPOD 지침이나 그 최신 확장판만으로는 완전히 다루기 어려운 고유한 도전 과제와 고려사항을 제시한다. 분류(classifier) AI 모델에서 생성형(generative) AI로의 전환이 이뤄지면서 이러한 특징이 더욱 부각되고 있다. 이에 이 논문에서는 이러한 충족되지 않은 요구를 해결하고, 빠르게 변화하는 연구현장에 유연하게 대응할 수 있도록 설계된 ‘TRIPOD-LLM statement’를 보고한다. 이 확장 지침은 원래 예측모델에 초점을 맞추었던 TRIPOD의 범위를 넘어, 진단부터 문서 요약까지 의료연구와 실무의 다양한 영역에 LLM이 미치는 광범위한 영향을 반영한다.
TRIPOD-LLM의 필요성
LLM은 자기회귀적(autoregressive) 구조를 가진 생성형 AI이다. 단순하게 말하면 앞선 단어들을 바탕으로 다음 단어를 예측하도록 학습된다는 의미이다. 그러나 이러한 기초적 학습만으로도 하나의 모델이 다양한 범위의 의료 관련 자연어 처리(natural language processing) 작업을 수행할 수 있다는 점이 확인되고 있다. 이러한 적응력은 주로 감독 학습 기반의 미세조정(supervised fine-tuning)이나 소수 예시 학습(few-shot learning) 방법을 통해 달성된다[
15,
16]. 이를 통해 LLM은 적은 예시만으로도 새로운 작업을 처리할 수 있다. 챗봇(chatbot) 솔루션(예: ChatGPT)은 LLM을 기반으로 하면서 두 가지 구성요소가 추가된다. 하나는 질의응답(질문에 대한 응답을 생성하는 instruction tuning 또는 supervised fine-tuning)이고, 다른 하나는 ‘얼라인먼트(alignment)’라고도 하는 선호도 랭킹이다. LLM과 챗봇에 특유한 이러한 방법론적 과정(예: 감독 미세조정에 사용된 하이퍼파라미터[hyperparameter]의 선택, 프롬프트[prompt] 설계의 복잡성, 모델 예측의 변동성, 자연어 출력 평가방법, 선호 기반 학습전략 등)은 현재의 보고 지침에서는 충분히 다루어지지 않고 있다. 이러한 과정은 모델의 신뢰성에 큰 영향을 미치므로, 별도의 구체적인 안내가 필요하다. 또한 LLM의 범용성과 생성형 특성은 기존 지침보다 더 세부적인 보고 지침을 요구한다. LLM은 특정 작업을 위해 훈련된 것이 아니며 훈련 데이터에 해당 작업이 반드시 포함되어 있지 않을 수 있으므로(기존 작업별 모델이 훈련 데이터에 해당 질병 유병률을 명시적으로 반영하는 경우와 달리), 신뢰성 있는 보고와 후속 안전성 확보를 위해서는 작업별로 특화된 지침이 필요하다.
생성형 출력물을 평가할 때 어떤 자동화된 혹은 인간 기반의 지표를 선택할 것인지에 대한 문제는 여전히 명확히 해결되지 않은 상태로, 현재 다양한 방법론이 적용되어 성능의 여러 측면을 평가하고 있다. 결과물이 완전히 비정형(unstructured) 텍스트이고 구조화된 라벨로 단순 환원이 불가능한 작업(예: 편지 생성, 요약 등)이라면 평가가 특히 복잡하다. 이런 경우 대부분의 자동 평가지표는 입력과 출력 텍스트 간의 중첩과 유사도에 초점을 맞추는데, 이는 생성된 텍스트의 사실적 정확성이나 적합성, 허위(hallucination)나 누락(omission)을 포착하지 못할 수 있다[
17-
19]. 이들 점수는 참조 텍스트와의 구조적ㆍ어휘적 유사성을 반영하지만, 이는 성능과 안전성을 종합적으로 평가하는 기준의 일부분에 불과하다. 인간의 텍스트 평가 또한 언어의 모호성, 임상 과제의 불확실성 등으로 인해 주관적일 수밖에 없다. 특히 의학 분야에서는 정답이 하나로 규정되지 않는 경우가 많고, 무작위적(aleatoric)이고 지식적(epistemic)인 불확실성이 모두 존재한다. 따라서 성능이 어떻게 평가되었는지를 보고하기 위한 구체적인 세부 지침이 필요하다. 본 논문에서는 LLM과 챗봇을 모두 포괄하여 ‘LLM’으로 지칭한다.
Table 1은 의료 분야에 적용 가능한 주요 작업유형을 정리하고, 관련 선행연구의 정의 및 예시를 제시한다[
5,
6,
20-
30].
LLM이 도입됨에 따라 환각(hallucination), 누락(omission), 신뢰성, 설명 가능성, 재현성, 프라이버시, 편향의 하위 전파 등과 같은 새로운 복잡성이 나타나고 있는데, 이는 임상 의사결정과 환자 진료에 부정적 영향을 미칠 수 있다[
20,
21,
31-
35]. 더불어 전자의무기록(electronic health record) 업체, 기술기업, 의료기관 간의 협력이 확대되면서, 실제 적용 일정은 현행 규제의 대응속도를 훨씬 앞지르고 있다[
8,
36]. LLM의 안전한 사용과 투명성 제고를 위해서는 개발 및 보고의 표준화가 필수적이다. 이는 일관성, 신뢰성, 검증 가능성 확보를 위한 것으로, 타 과학 분야에서 확립된 임상평가와 유사한 수준의 기준이 필요하다[
37-
39].
방법
TRIPOD-LLM 지침은 LLM을 개발, 튜닝 또는 평가하는 모든 의료 응용 또는 맥락에서 연구 보고를 안내하기 위해 작성되었으며, 기존 TRIPOD 지침 개발과정과 동일한 절차를 따랐다. 본 분야에 시의적절한 보고 지침이 필요하다는 점을 고려하여 신속한 델파이(Delphi) 과정을 적용하였고, 이를 생명력 있는(living) 지침 방식과 결합하였다. 관련 용어에 대한 정의는 부록(
Box 1)으로 수록하였다.
가이드라인 개발을 위한 운영위원회를 구성하고, natural language processing (NLP), AI, 의료정보학 등 다양한 전문성과 경험을 갖춘 전문가 패널과 함께 작업하였다. (본 논문 저자의 두 그룹 내 역할은 Author contributions에 기술했다.) 이 지침은 2024년 5월 2일, EQUATOR Network에 개발 중인 보고 지침으로 등록되었다(
https://www.equator-network.org).
윤리 선언
본 연구는 2024년 3월 26일 MIT 인간대상 실험 윤리위원회(COUHES IRB)로부터 면제 승인을 받았다(exempt ID: E-5705). 델파이 설문 참여자는 설문 응답 전 전자 동의서를 제출하였다.
후보 항목 목록 도출
TRIPOD-2015, TRIPOD+AI 가이드라인(
https://www.tripod-statement.org) 및 LLM 보고 지침 관련 문헌을 참고하여 초기 후보 항목 목록을 작성하였다[
9,
11,
37,
40]. 운영위원회와 전문가 패널은 추가 문헌조사를 통해 이 목록을 확장하였고, 최종적으로 제목, 초록, 서론, 방법, 결과, 논의, 기타 항목 등 총 64개의 고유 항목으로 표준화하였다.
패널 모집
델파이 참여자는 운영위원회가 관련 논문 저자와 개인 추천, 그리고 델파이 참여자가 추천한 전문가를 포함하여 선별하였다. 운영위원회는 지리적 및 학문적 다양성을 반영하여 연구자(통계학자, 데이터 과학자, 역학자, 머신러닝 전문가, 임상의, 윤리학자), 의료전문가, 학술지 편집자, 연구비 지원자, 정책 입안자, 의료규제자, 환자 옹호단체 등 주요 이해관계자를 포함시켰다. 참여자 수에는 최소 표본크기 제한을 두지 않았다. 운영위원회 구성원이 각 참여자의 전문성 또는 경험을 확인하였다. 이후 이메일을 통해 설문 참여를 요청하였으며, 참여자에게 금전적 보상이나 선물은 제공하지 않았다.
델파이 과정
설문은 개별 응답이 가능하도록 영어로 설계되었으며, Google Forms (Google LLC)를 통해 온라인으로 배포되었다. 모든 응답은 익명으로 처리하였고, 이메일 또는 식별 정보는 수집하지 않았다. 참여자에게 각 항목을 ‘생략 가능,’ ‘포함 가능,’ ‘포함 권장,’ ‘포함 필수’로 평가하도록 요청하였다. 이는 이전 TRIPOD 가이드라인에도 적용한 방식이다[
9]. 모든 항목에 대해 의견을 남기거나 새로운 항목을 제안할 수 있었다. 자유 응답은 D.S.B.와 J.G.가 수합 및 분석하였으며, 그 결과를 토대로 항목의 문구 수정, 통합, 신규 항목 제안을 진행하였다. 모든 운영위원회 구성원도 델파이 설문에 참여할 수 있도록 초대되었다.
1차 참여자
1차 설문은 2024년 3월 1일부터 4월 23일까지 실시하였고, 설문 링크는 56명에게 발송하였다. 이 중 26명이 설문을 완료하였다. 설문 참여자는 9개국 출신으로, 북미 14명, 유럽 5명, 아시아 2명, 남미 1명, 오스트랄라시아 1명이었다. 3명은 정보를 제공하지 않았다. 참여자는 주된 업무 분야를 복수로 선택할 수 있었는데, 26명 중 20명(77%)이 AI, 머신러닝, 임상정보학, NLP 분야를, 14명이 의료 분야를 주요 분야로 선택하였다.
합의 회의
4월 22일과 24일, D.S.B.와 J.G.의 주재하에 모든 운영위원회 및 전문가 패널 구성원을 초대하여 온라인(Zoom; Zoom Video Communications Inc.)으로 합의 회의를 진행하였다. 회의 녹화본과 회의록은 불참자를 위해 즉시 배포하여 회의 이후에도 의견 제출이 가능하도록 하였다. 각 질문에 대한 응답과 자유 의견을 차례로 검토하였고, ‘포함 필수’에 대해 50% 미만의 지지를 받은 항목은 따로 표시하여 포함의 중요성에 대해 심도 있게 논의하였다. 모든 항목에서 합의(consensus)가 이뤄졌으며, 제3자의 개입은 필요하지 않았다. 합의에 도달할 때까지, 패널 중 추가 의견이나 이견이 없을 때까지 항목을 논의하였다. 논의 녹취록은 개인식별 정보와 민감 정보를 제거한 후 Supplementary Information에 공개하여 투명성을 확보하였다.
LLM 활용 분야의 방대함을 고려하여, ‘연구설계(Research Design)’와 ‘LLM 과업(LLM Task)’ 아래 추가 하위 범주로 항목을 그룹화하는 모듈형 방식을 도입하였다. 이 방안은 합의 회의에서 채택되었고, 최종 분류는 운영위원회의 승인을 받았다.
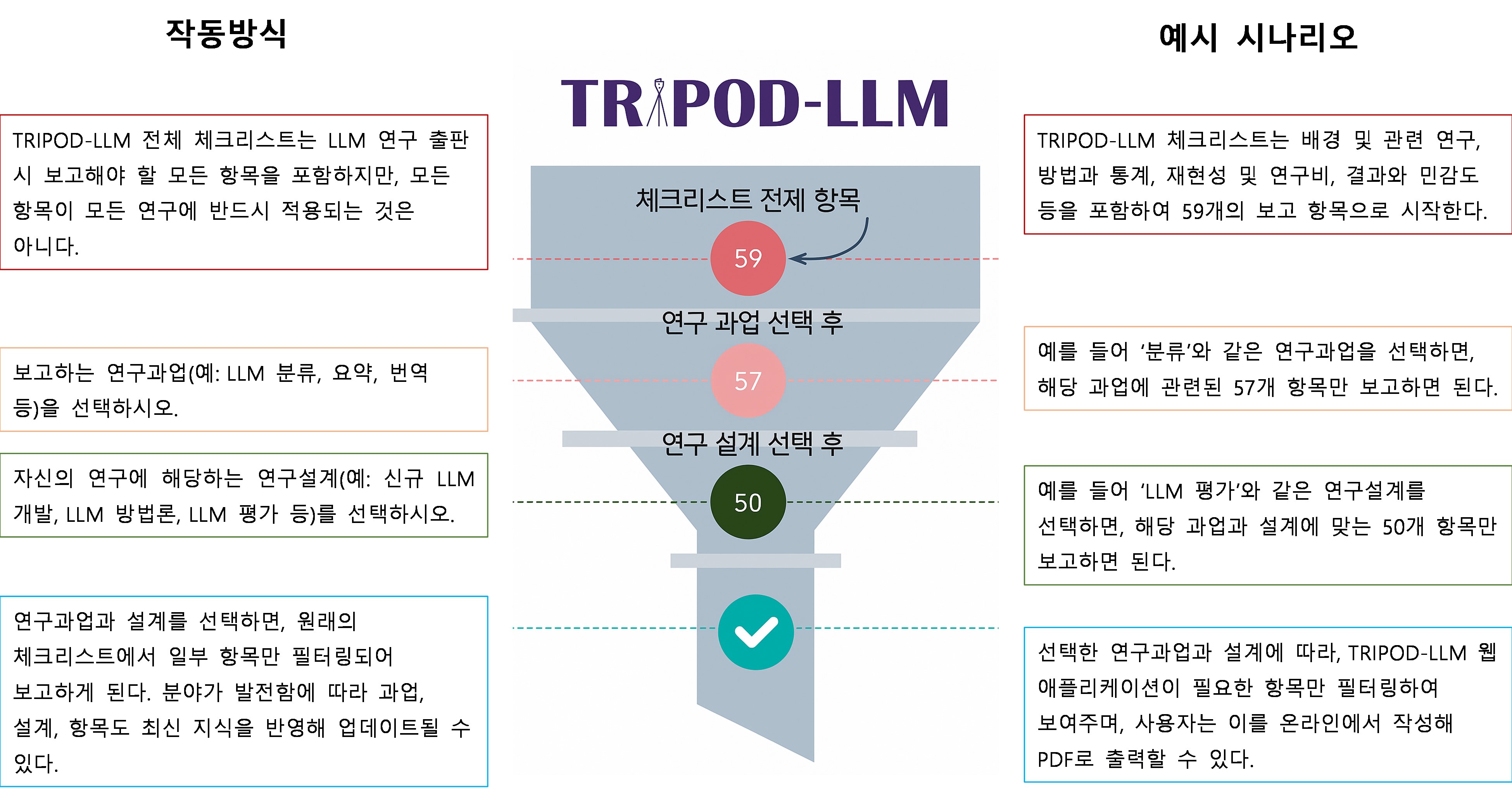
LLM의 개발, 튜닝, 평가, 적용 등 다양한 단계와 여러 의료과업에서의 활용을 적절히 반영하기 위해, 항목은 (1) 연구설계와 (2) LLM 과업을 기준으로 분류하였다(
Fig. 1). 연구설계 범주는
de novo LLM 개발, LLM 방법(미세조정, 프롬프트 엔지니어링, 아키텍처 수정 등), LLM 내재 평가, 헬스케어 환경에서의 LLM 평가 등으로 구분된다. LLM 과업 범주는 저수준 텍스트 처리(품사 태깅, 관계 추출, 명명 엔티티 인식 등), 분류(진단 등), 장문의 질의응답, 대화형 에이전트, 문서 생성, 요약/단순화, 기계번역, 결과 예측(예: 예후 등)이다. 각 항목은 복수의 설계 및 과업 범주에 해당할 수 있고, 한 연구에 둘 이상의 설계 및 과업이 포함될 수도 있다. 연구에 포함된 모든 설계 및 과업에 해당하는 항목은 반드시 보고해야 한다. 각 설계 및 과업 범주에 대한 정의와 예시는
Table 1에 제시하였다. 이러한 분류가 완벽하지 않으며, 설계 및 과업 간 중복이 존재할 수 있음을 인정한다.
TRIPOD-LLM 지침
TRIPOD-LLM은 LLM을 개발, 튜닝, 프롬프트 엔지니어링하거나 평가하는 연구에서 필수적인 항목들을 적절하게 보고할 수 있도록 구성된 체크리스트를 포함한다(
Table 2).
Box 2에는 TRIPOD-2015 및 TRIPOD+AI에 추가되거나 변경된 주요 내용이 요약되어 있으며,
Box 1에는 주요 정의가 제시되어 있다. TRIPOD-LLM 체크리스트는 제목(1개 항목), 초록(1개 항목), 서론(2개 항목), 방법(8개 항목), 오픈 사이언스 실천(1개 항목), 환자 및 대중의 참여(1개 항목), 결과(3개 항목), 논의(2개 항목) 등 총 19개 주요 항목으로 구성되어 있다. 이 주요 항목들은 50개의 세부 항목으로 세분화된다. 이 중 14개 주요 항목과 32개 세부 항목은 모든 연구설계와 LLM 작업에 공통으로 적용되고, 나머지 5개 주요 항목과 18개 세부 항목은 특정 연구설계나 LLM 작업유형에만 적용된다. 방법(Methods)에서 논의된 바와 같이, TRIPOD-LLM 지침은 다양한 LLM 연구유형에 대응할 수 있도록 모듈형 형식을 도입하였다(
Table 1). 일부 항목은 특정 연구설계나 LLM 작업유형에만 해당된다. 이러한 설계 및 작업 범주는 폭넓지만 상호 배타적인 것은 아니며, 연구의 맥락에 따라 달라질 수 있고 LLM의 적용이 진화함에 따라 변화가 필요할 수 있다. 또한 LLM 기반 연구의 학술지 또는 학회 초록을 위한 별도의 체크리스트를 포함하고 있으며, 기존 TRIPOD+AI for abstracts 지침도 개정되어(TRIPOD-LLM for abstracts) (
Table 3), 새로운 내용을 반영하고 TRIPOD-LLM과의 일관성을 유지하였다[
18].
TRIPOD-LLM에 포함된 권고사항은 LLM 기반 연구가 어떻게 수행되었는지 완전하고 투명하게 보고하기 위한 것으로, LLM을 개발하거나 평가하는 방법 자체를 규정하지는 않는다. 이 체크리스트는 연구의 질을 평가하는 도구가 아니며, CANGARU (ChatGPT, generative artificial intelligence and natural large language models for accountable reporting and use) [
41] 및 CHART (Chatbot Assessment Reporting Tool) [
40] 역시 생성형 AI와 챗봇에 초점을 맞춘 보완적 지침이다.
임상 및 병원 운영작업을 위해 LLM의 사전학습(pretraining), 미세조정(fine-tuning), 후향 평가(retrospective evaluation), 임상 적용(clinical deployment)에 관한 이전에 발표된 연구를 대상으로 작성한 TRIPOD-LLM 체크리스트의 완성 예시는
Supplement 1에 제시되어 있다[
6]. 또한 이해를 돕기 위해 작성용 체크리스트는
Supplement 2으로, 새로운 항목에 대한 설명과 보충 문서는
Supplement 3으로 제공하였다.
생명력 있는(living) 문서로서의 TRIPOD-LLM 지침
이 분야의 급속한 발전속도와 의료종사자 및 환자와의 상호작용 시점을 고려하여, (생명)과학 및 기타 의료 분야에서 LLM을 적시에 활용할 수 있도록 TRIPOD-LLM 지침을 신속히 마련하기로 결정하였다. 이 지침은 사용자 테스트를 통한 개선, 분야 발전에 따른 업데이트, 새로운 표준의 도입 및 정기적인 검토를 용이하게 하기 위하여, 생명력 있는(living) 문서형태로 설계되어 인터랙티브 웹사이트에 게시되어 있다. 따라서 향후 보고 권고사항의 지속적인 변화가 예상되므로, 사용자들은 항상
https://tripod-llm.vercel.app/에 게시된 최신 버전의 가이드라인을 참고하는 것이 좋다.
TRIPOD-LLM 지침을 유연한 변화가 가능한(living) 방식으로 개발한 것은, 변화하는 근거에 기반하여 최신 권고를 제공하기 위해 고안된 living systematic reviews [
42,
43] 및 임상진료지침(clinical practice guidelines) [
44,
45] 개발의 경험에서 영감을 받은 것이다. 지침에 대한 공공 의견은, 접근성을 높이기 위해 여러 경로—프로젝트별 GitHub 저장소, TRIPOD-LLM 웹사이트, 메인 TRIPOD 웹사이트(
https://www.tripod-statement.org/)—를 통해 수집할 예정이다. 모호하거나 중복된 표현 등 지침의 가독성과 내용 전반에 대한 의견 모두를 환영한다. 예를 들어, 사용자는 실제 적용 가능성을 높이기 위한 항목 변경, 새로운 항목 추가, 특정 연구설계 또는 LLM 작업 모듈에 배정된 항목 추가/삭제, 연구설계 또는 LLM 작업 모듈 범주 변경 등을 제안할 수 있다.
전문가 패널은 3개월마다 회의를 열어 업데이트를 논의한다. 회의 전, 패널 구성원들은 그동안의 주요 문헌을 검토하여 업데이트에 참고한다. 업데이트 단위는 지침에 명시된 체크리스트 항목, 연구설계 범주, LLM 작업 범주가 된다. 회의에서 패널은 현재 지침의 상태를 점검하고, 공공 의견, 문헌 검토, 주제 전문성을 고려하여 개정사항을 제안한다. 운영위원회는 논의내용을 반영해 지침을 수정하고, 이를 전문가 패널에 회람하여 최종 검토 및 승인을 받는다. 검토 결과에 따라 TRIPOD-LLM 지침의 각 구성요소(항목, 연구설계, LLM 작업)에는 다음과 같은 조치가 취해질 수 있다[
42]: (1) 변경 없음; (2) 실질적 내용의 수정(명확성을 위한 간단한 문구 수정이나 오ㆍ탈자 교정 등은 수정에 해당하지 않음); (3) 하나 이상의 구성요소를 통합(통합은 동일한 구성요소 유형 내에서만 이루어짐); (4) 하나의 구성요소를 둘 이상으로 분리(분리 역시 동일한 구성요소 유형 내에서만 적용); (5) 해당 구성요소를 지침에서 삭제.
고찰
TRIPOD-LLM은 생물의학 및 의료 분야에서 급속하게 발전하고 있는 LLM 분야에서 연구자와 학술지, 의료전문가, 상업적ㆍ비상업적 LLM 개발자, 그리고 의료기관을 위한 안내를 목적으로 개발되었다. 이 지침은 LLM의 개발, 튜닝, 평가를 다루는 연구에 대한 최소한의 보고 권고사항을 제시한다. TRIPOD-LLM 항목에 따라 보고함으로써 연구자는 LLM 연구방법의 질을 이해하고 평가할 수 있다. 또한 연구결과의 투명성을 높이고, 연구결과의 과해석을 줄이고, 재현성과 복제를 용이하게 하며, LLM의 실제 적용에도 도움이 된다.
모델 생애주기 전반에 걸친 투명성은 본 가이드라인에서 강하게 강조되는 핵심 요소이다. LLM 생애주기 각 단계에서의 세부적인 문서화가 요구된다[
46]. 예를 들어, 개발 및 미세조정 단계에서는 학습 데이터의 출처와 전처리(preprocessing) 과정을 공개하는 것이 중요하다. 또한 LLM의 버전과 기존의 파운데이션 모델에 대한 미세조정 또는 얼라인먼트 과정에 대한 세부 사항을 투명하게 보고해야 LLM 간의 공정한 비교가 가능하다. 여기에는 학습 데이터 수집 시점의 기준일(cut-off date)을 명시하여, 데이터 세트의 시간적 적합성과 평가 시의 데이터 유출 또는 오염 가능성을 분명히 하는 내용이 포함된다. 아울러, 연구에서는 모델 버전의 날짜, 데이터 수집 중 모델이 고정(frozen)되어 있었는지 혹은 동적으로 업데이트되었는지도 반드시 기록해야 한다. 입력 데이터의 투명성 역시 필수적이다. LLM은 보통 여러 공개 대규모 데이터 세트로 훈련되므로, 사회적 편향이나 불평등(낙인을 찍는 용어, 집단 간 통계적 위험 배분 등)이 내재화될 위험이 있다. 따라서 데이터 소스의 선별과 잠재적 편향에 대한 체계적이고 투명한 접근이 요구된다[
20,
32,
47-
50].
TRIPOD-LLM 지침은 인간의 통찰과 감독(human insight and oversight)도 중요한 구성요소로 다루며, 이는 LLM의 책임감 있는 실제 적용을 위해 필수적인 요소이다(다만, 배포 신뢰성과 관찰 가능성 자체는 본 논문의 범위를 벗어난다)[
51-
53]. 본 가이드라인은 LLM의 예상 적용 맥락에 대한 보고를 강화하고, 해당되는 경우 LLM에 부여된 자율성 수준을 명확히 보고할 것을 요구한다. 또한 데이터 세트 구축과 평가 시의 품질관리 절차(예: 평가자 자격, 이중 평가 요구사항, 평가자에게 제공되는 구체적 지침 등)도 강조하며, 이를 통해 텍스트 평가의 미묘한 부분까지 포착하여 안전성과 성능에 대한 신뢰성 있는 평가가 가능하도록 한다.
프롬프트와 과업 특이적 성과 보고는 LLM의 고유 특성으로 인해 필수적으로 추가된 항목이다. 프롬프트 엔지니어링 방법의 차이는 LLM 성능에 큰 영향을 미칠 수 있고, 이는 벤치마크 비교나 실제 적용 가능성을 왜곡할 위험이 있다[
54,
55]. 따라서 관련이 있는 경우라면 프롬프트 개발에 사용된 데이터 소스, LLM 모델명과 버전, 수행된 전처리 단계, 프롬프트 엔지니어링 방법을 모두 상세히 기술해야 한다. 이를 통해 프롬프트가 LLM으로부터 안정적이고 재현 가능한 성능을 이끌어내도록 설계되었는지 확인할 수 있다. 추가로, 평가 환경(지침, 인터페이스, 평가에 참여한 집단의 특성 등)에 대한 명확한 보고도 요구된다. 이는 LLM의 성능이 실제 적용 환경과 유사한 조건에서 평가되었는지를 강조하여 그 실용적 유용성을 측정하는 믿을 만한 기준으로 삼기 위함이다.
TRIPOD-LLM의 주요 사용자 및 수혜자는 (1) 논문을 저술하는 학계 및 산업계 연구자, (2) 연구 논문을 평가하는 학술지 편집자 및 심사위원, (3) 투명성과 연구 질 제고의 혜택을 받을 기타 이해관계자(연구 공동체, 학술기관, 정책 입안자, 연구비 지원기관, 규제기관, 환자, 연구 참여자, 산업계, 일반 대중 등)가 될 것이다. 편집자, 출판사, 산업계에서는 학술지 저자 지침 내에 TRIPOD-LLM 링크를 명시하고, 논문 제출 및 심사과정에서 활용을 권고하며, 권고사항 준수를 표준으로 삼을 것을 권장한다. 또한 연구비 지원기관에서도 LLM 연구 지원 신청 시 TRIPOD-LLM에 따라 보고계획을 포함하도록 요구하여, 연구의 낭비를 줄이고 연구비의 효용을 높일 수 있을 것이다.
이 가이드라인은 주로 텍스트 전용 LLM을 염두에 두고 개발되었으나, 최근 LLM이 통합된 멀티모달(multi-modal) 모델(예: 비전-언어 모델[
56])도 빠르게 등장하고 있어, 신속하고 유연한 보고 지침이 특히 필요하다. 보고 고려사항의 상당수는 텍스트 LLM과 멀티모달 모델 모두에 적용되지만, 예를 들어 비전-언어 모델에서는 텍스트와 이미지의 전처리 과정을 모두 보고해야 한다. 그러나 향후 버전에는 멀티모달 특유의 고려사항도 반영할 필요가 있다. 예컨대, 영상 데이터를 활용하는 LLM 개발 연구는 영상 획득에 대한 세부 정보를 보고해야 한다. 당분간 LLM을 주된 구성요소로 포함하는 방법의 개발/평가 연구는 TRIPOD-LLM 지침을 사용할 것을 권고하지만, 이 부분은 해석에 따라 다를 수 있음을 인정한다. 사용자는 재현성과 이해 가능성, 투명성이라는 목표를 염두에 두고, 상식에 기반해 적절한 보고 지침을 선택하고 TRIPOD-LLM 항목 중 멀티모달 LLM에 적용할 수 있는 요소를 해석하여 보고해야 한다. 필요 시 방사선학(radiomics) 등 AI 타 분야의 방법론 가이드라인도 참고할 수 있다[
57,
58].
임상 AI의 모델 메타데이터를 요약하는 model card의 생성, 검증, 인증, 유지ㆍ관리에는 Coalition for Health AI [
59], Epic AI Labs [
60,
61]와 같은 검증 연구소나 내부 검증기준이 중요한 역할을 하게 될 것이다. TRIPOD-LLM 기준은 이러한 연구소들이 규제기준에 부합하는 LLM 검증방안을 마련하고(예: 미국 바이든 행정부의 ‘안전하고 신뢰할 수 있는 인공지능에 관한 행정명령,’ 미국 AI Safety Institute [
62], 미국 ONC HTI-1 Final Rule [
63], EU AI Act [
64]), 임상 AI의 신뢰성과 유용성에 대해 환자와 임상의, 그리고 이해관계자의 신뢰를 형성하는 데 기여할 것으로 기대된다.
LLM을 평가하고 검증하려면 특수한 전문성과 자원이 필요하다는 점도 강조할 필요가 있다. LLM을 공정하고 안전하게 도입하려면 개발에 대한 투자뿐 아니라, 대형 학술기관 외의 환경에서도 견고한 검증이 가능하도록 하는 인프라 구축에 대한 투자도 병행되어야 한다. LLM이 시간과 지역에 따라 변화하는 맥락을 내포하고 있으므로 모델의 성능과 공정성이 시점이나 기관에 따라 달라질 수 있다는 점을 고려할 때, 이 체크리스트는 LLM 평가를 위한 지속적인 과정의 일부로 인식되어야 한다. 사용자에 따라 결과가 달라지는 LLM의 특성으로 인해 기존 ML 모델보다 변화 양상이 더욱 예측하기 어려울 수 있으므로, 단일 시점의 검증 결과만으로 보편적 유효성을 주장하기보다는 효과의 경향성과 이질성을 파악하는 데 더 집중해야 한다.
현재 TRIPOD-LLM 체크리스트의 한계는, 이 분야의 전례 없이 빠른 개발 및 출판 속도로 인해 연구 공동체를 위한 신속한 가이드라인 개발이 불가피했다는 점에서 비롯된다. 본 연구에서는 초기 체크리스트 도출을 위해 신속한 델파이 과정을 거쳤으나, 이로 인해 합의와 입력의 폭이 제한될 수 있었음을 인정한다. 이에 따라, 피드백을 신속하게 반영하고 변화하는 방법론에 적응할 수 있도록 생명력 있는(living) 지침방식을 도입하였다. 이러한 특성상 본 논문에 포함된 체크리스트는 시간이 지나면 구 버전이 될 수 있으므로, 사용자는 항상
https://tripod-llm.vercel.app/에서 최신 버전을 확인해야 한다.
결론
TRIPOD-LLM은 연구자가 자신의 연구를 완전하게 보고할 수 있도록 돕고, LLM 개발자와 연구자, 전문가 심사자(peer reviewer), 편집자, 정책 입안자, 최종 사용자(예: 의료전문가), 그리고 환자가 LLM 기반 연구의 데이터, 방법, 결과, 결론을 명확히 이해할 수 있도록 지원하는 것을 목표로 한다. TRIPOD-LLM 보고 권고사항을 준수하면 연구에 소요되는 시간과 노력, 비용을 가장 효율적으로 활용할 수 있으며, LLM 연구의 가치를 높이고 긍정적 영향을 극대화할 수 있을 것이다.
온라인 콘텐츠
-
Authors’ contributions
DSB, JG, LAC, GSC, and KGMM were on the steering group that directed the guideline-development process. SC, CF, DR, GS, TM, DDF, RU, LHH, YA, JWG, LGM, NM, and RD were members of the expert panel. DSB and JG drafted the initial list of candidate items.
These authors contributed equally: Majid Afshar, Saleem Ameen, Yindalon Aphinyanaphongs, Shan Chen, Giovanni Cacciamani, Dina Demner-Fushman, Dmitriy Dligach, Roxana Daneshjou, Chrystinne Fernandes, Lasse Hyldig Hansen, Adam Landman, Lisa Lehmann, Liam G. McCoy, Timothy Miller, Amy Moreno, Nikolaj Munch, David Restrepo, Guergana Savova, Renato Umeton, and Judy Wawira Gichoya.
-
Conflict of interest
DSB is an associate editor at Radiation Oncology and HemOnc.org, receives research funding from the American Association for Cancer Research, and provides advisory and consulting services for Mercurial AI. DDF is an associate editor at the Journal of the American Medical Informatics Association, is a member of the editorial board of Scientific Data, and receives funding from the intramural research program at the US National Library of Medicine, NIH. JWG is a member of the editorial board of Radiology: Artificial Intelligence, BJR Artificial Intelligence, and NEJM AI. All other authors declare no potential conflict of interest relevant to this article.
-
Funding
JG is funded by the US National Institutes of Health (NIH) (U54 TW012043-01 and OT2OD032701). SC is supported by the NIH (U54CA274516-01A1). GSC is supported by Cancer Research UK (program grant C49297/A27294) and by the Engineering and Physical Sciences Research Council grant for ‘Artificial intelligence innovation to accelerate health research’ (EP/Y018516/1), and is a National Institute for Health and Care Research (NIHR) senior investigator. The views expressed in this article are those of the author and not necessarily those of the NIHR or the Department of Health and Social Care. YA is partially supported by the NIH (3UL1TR001445-05) and the National Science Foundation Award (1928614 and 2129076). LAC is funded by the NIH (U54 TW012043-01, OT2OD032701 and R01EB017205). DSB is supported by the NIH (U54CA274516-01A1 and R01CA294033-01) and the American Cancer Society and American Society for Radiation Oncology grant ASTRO-CSDG-24-1244514-01-CTPS. JWG receives support from 2022 Robert Wood Johnson Foundation Harold Amos Medical Faculty Development Program, a Radiological Society of North America Health Disparities grant (EIHD2204), the Lacuna Fund (67), the Gordon and Betty Moore Foundation, an NIH (National Institute of Biomedical Imaging and Bioengineering) MIDRC grant under contracts 75N92020C00008 and 75N92020C00021, and an NHLBI award (R01HL167811).
-
Data availability
Research data are available at https://doi.org/10.1038/s41591-024-03425-5
-
Acknowledgments
None.
Supplementary materials
Fig. 1.TRIPOD-LLM 워크플로우. TRIPOD-LLM 체크리스트 워크플로우는 총 59개의 보고 항목으로 시작하며, 연구과업(예: 분류, 요약)과 연구설계(예: LLM 평가) 선택에 따라 필요한 항목 수가 점차 줄어든다. 두 가지 모두 선택한 후에는, 보고에 필요한 항목만 필터링된 목록이 생성된다. TRIPOD, transparent reporting of a multivariable model for individual prognosis or diagnosis; LLM, large language model.
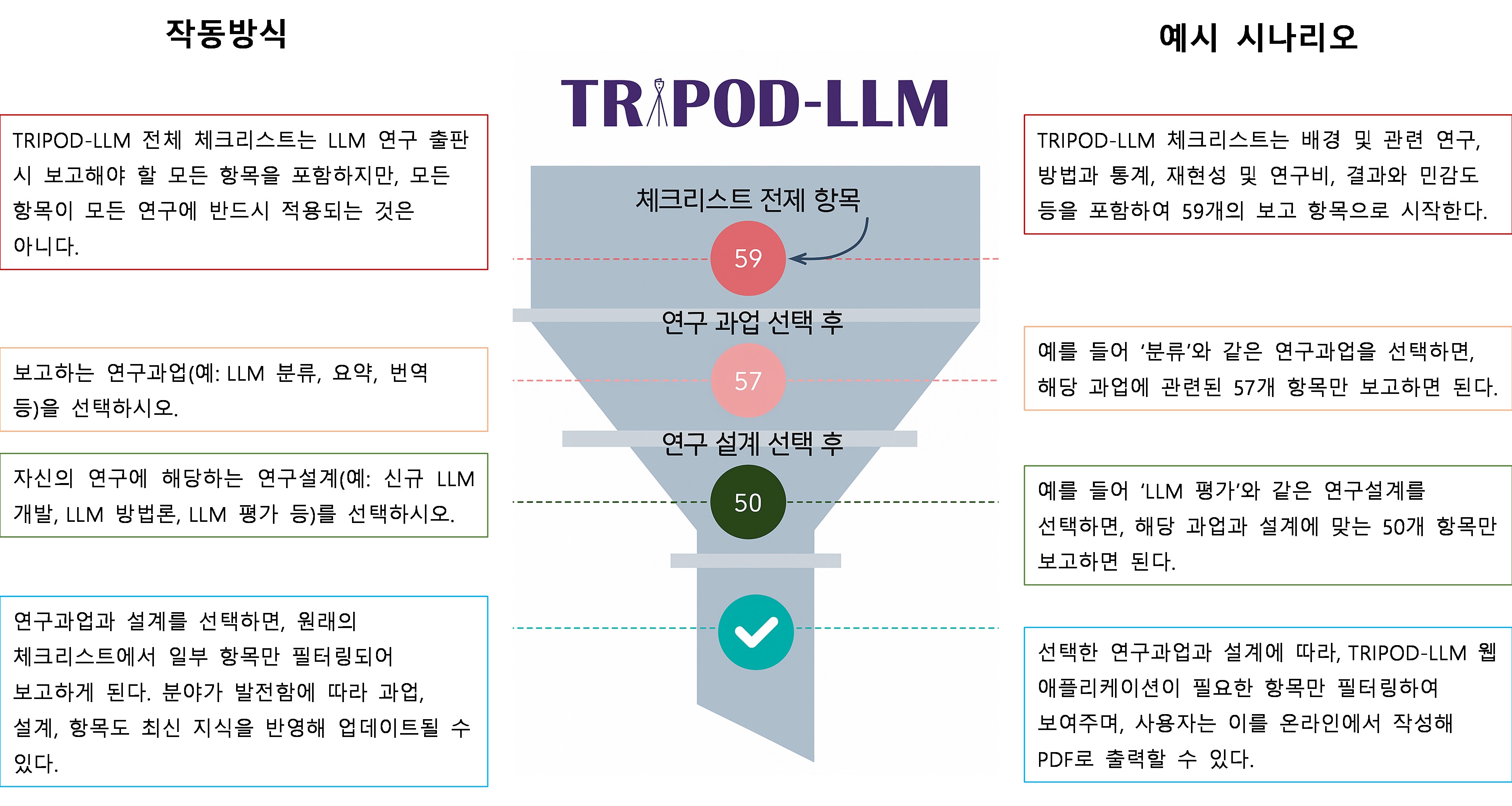
Table 1.TRIPOD-LLM 가이드라인의 모듈형 연구설계 및 LLM 과업 범주
|
과업(task) |
정의(definition) |
예시(example) |
|
연구 설계(research design) |
|
|
|
De novo LLM 개발 |
새로운 언어 모델을 처음부터 구축하거나, 기존 기본(base) 모델을 상당 부분 미세 조정하여 새로운 기능을 개발하거나 새로운 작업에 적응시키는 작업 |
병원 임상 데이터로 새로운 LLM을 사전 학습(pretraining)하는 연구[22] |
|
LLM 방법(LLM methods) |
언어 모델의 새로운 아키텍처, LLM을 이해하기 위한 새로운 계산방법, LLM 평가를 위한 새로운 방법, LLM 프롬프트 최적화를 위한 새로운 방법 등에 중점을 두는 정량적 또는 이론적 연구 |
의료 분야에서 retrieval-augmented generation LLM 프레임워크를 연구하는 연구[23] |
|
LLM 평가(LLM evaluation) |
기존 LLM의 효율성, 정확성, 특정 의료작업에의 적합성을 평가하거나, 사용 시 발생하는 위험과 편향을 평가하는 작업 |
기존 LLM에서 편향된 진단추론을 조사하는 연구[20] |
|
의료환경에서의 LLM 평가(LLM evaluation in healthcare settings) |
임상 워크플로우 내에서 LLM이 통합되어 실제로 사용될 때, 임상적ㆍ행정적ㆍ인력 관련 결과(outcomes) 측면의 영향과 통합에 초점을 두고 평가하는 작업 |
병원 입원환자에서 실시간(real-time) 예후예측을 위해 LLM을 배치하여 그 성능을 보고하는 연구[6] |
|
LLM 과업(LLM task) |
|
|
|
텍스트 처리(text processing) |
토큰화(tokenization), 구문 분석(parsing), 엔터티 인식(entity recognition) 등, 텍스트 데이터를 조작하거나 하위 수준으로 처리하는 작업을 포함하되, 이에 국한되지는 않음 |
LLM 기반 명명 엔터티 인식(named entity recognition) 방법을 연구하는 연구[24] |
|
분류(classification) |
텍스트 데이터에 미리 정의된 라벨을 할당하는 작업 |
임상노트에서 1개 이상의 사회적 결정요인(social determinants of health)이 언급되었는지 여부를 판단하기 위해 LLM을 미세 조정하는 연구[25] |
|
장문 질의응답(long-form question answering) |
복잡한 질의에 대해 여러 문서 또는 근거를 바탕으로 상세한 답변을 제공하는 작업(객관식 질의응답[multichoice Q&A]은 ‘분류[classification]’에 포함됨) |
기존 LLM이 환자 포털 메시지에 답변하는 능력을 조사하는 연구[21] |
|
정보 검색(information retrieval) |
대규모 데이터 세트에서 특정 질의에 따라 관련 정보를 추출하는 작업. 문헌 리뷰, 환자 병력 조회 등에 적용됨 |
트랜스포머 모델을 훈련하여 사용자의 질의에 적합한 생의학 논문을 검색하도록 한 연구[26] |
|
대화형 에이전트(conversational agent, 챗봇) |
사용자와 대화를 이어가는 작업. 환자 상호작용, 건강상담, 의료진의 가상 비서 등으로 활용됨 |
LLM 기반 챗봇 접근 가능 여부가 임상의의 진단추론에 미치는 영향을 조사하는 연구[27] |
|
문서 생성(documentation generation) |
임상 데이터, 녹취, 기록 등을 바탕으로 자동으로 의료문서를 생성하는 작업 |
임상환경 녹음에서 자동 생성된 임상노트의 품질을 평가하는 연구[5] |
|
요약 및 단순화(summarization and simplification) |
방대한 텍스트 문서를 요약하거나, 쉽게 이해할 수 있도록 내용을 단순화하는 작업. 환자 교육, 의무기록 요약 등에서 유용함 |
LLM이 퇴원 요약(discharge summary)을 환자 친화적 평이문으로 변환하는 능력을 평가하는 연구[28] |
|
기계 번역(machine translation) |
한 언어의 텍스트를 다른 언어로 변환하는 작업 |
번역에 특화된 소형 언어모델과 범용 LLM이 스페인어-영어 생의학 텍스트를 번역하는 능력을 비교한 연구[29] |
|
결과 예측(outcome forecasting) |
과거 데이터를 기반으로 미래의 의료결과를 예측하는 작업. 예후 평가나 치료효과 연구에 활용됨 |
LLM이 중환자실 입원 환자의 병원 외 사망률을 예측하는 능력을 연구한 논문[30] |
Table 2.
|
섹션(section) |
항목(item) |
설명(description) |
연구설계(research design) |
LLM 과업(LLM task) |
|
제목(Title) |
1 |
해당 연구가 LLM의 개발, 미세조정 또는 성능 평가임을 밝히고, 수행작업, 대상 집단, 예측하고자 하는 결과를 명시할 것 |
All |
All |
|
초록(Abstract) |
2 |
TRIPOD-LLM for abstracts 참조 |
All |
All |
|
서론(Introduction) |
|
|
|
|
|
배경(Background) |
3a |
의료 맥락/활용 사례(예: 행정, 진단, 치료, 임상 워크플로우)와 LLM 개발 또는 평가의 근거를 설명하고, 기존 접근법 및 모델을 인용할 것 |
All |
All |
|
3b |
대상 집단과 케어 경로 내에서 LLM의 의도된 사용, 현재 표준 실무의 의도된 사용자(예: 의료전문가, 환자, 대중, 행정가)를 포함하여 설명할 것 |
E, H |
All |
|
목적(Objectives) |
4 |
연구목적을 명시하되, 연구가 LLM의 초기 개발, 미세조정, 검증 중 어떤 단계(혹은 여러 단계)에 대한 것인지 포함할 것 |
All |
All |
|
방법(Methods) |
|
|
|
|
|
데이터(Data) |
5a |
학습, 튜닝, 평가 데이터 세트의 출처를 개별적으로 기술하고, 해당 데이터를 사용한 근거를 제시할 것(예: 웹 코퍼스[corpus], 임상연구/시험 데이터, EHR 데이터 등) |
All |
All |
|
5b |
관련 데이터 항목을 기술하고, 분포 등 데이터 세트의 정량적ㆍ정성적 특성과 기타 관련 설명자(예: 출처, 언어, 국가 등) 제공 |
All |
All |
|
5c |
개발(학습, 미세조정, reward modeling) 및 평가 데이터 세트에서 사용한 텍스트의 가장 오래된 날짜와 최신 날짜를 명확히 제시할 것 |
All |
All |
|
5d |
데이터 전처리 및 품질 검사방법을 기술하되, 이 과정이 텍스트 코퍼스, 기관, 사회인구학적 집단 간에 동일했는지 포함할 것 |
All |
All |
|
5e |
결측 및 불균형 데이터 처리방법과 데이터 제외 사유를 명확히 기술할 것 |
All |
All |
|
분석방법(Analytical methods) |
6a |
LLM 이름, 버전, 최종 학습날짜를 보고할 것 |
All |
All |
|
6b |
LLM 개발과정(아키텍처, 학습, 미세조정 절차, 얼라인먼트 전략[예: 강화학습, 직접 선호도 최적화]과 그 목표[예: 유용성, 정직성, 무해성 등])에 대한 세부사항 보고 |
M, D |
All |
|
6c |
LLM을 활용한 텍스트 생성과정, 프롬프트 엔지니어링(출력 일관성 포함), 추론 설정(예: 시드 값, 온도, 최대 토큰 길이, 페널티 등) 상세 보고 |
M, D, E |
All |
|
6d |
LLM의 초기 및 후처리 출력값 명시(예: 확률, 분류, 비정형 텍스트 등) |
All |
All |
|
6e |
분류(classification) 관련 세부 내용 및 확률 산출방법과 임계값 식별방법 포함 시 상세 설명 |
All |
C, OF |
|
LLM 출력(LLM output) |
7a |
생성결과의 품질(일관성, 관련성 등) 평가지표 포함 |
All |
QA, IR, DG, SS, MT |
|
7b |
배포 시점의 하위 과업 결과지표의 적합성과, 해당 용도와 인간 평가와의 상관관계 보고 |
E, H |
All |
|
7c |
결과 정의, LLM 예측 산출방식(예: 공식, 코드, 객체, API), 폐쇄형 LLM의 추론날짜, 평가지표 명확화 |
E, H |
All |
|
7d |
결과 평가에 주관적 해석이 필요할 경우, 평가자의 자격, 제공된 지침, 평가자 인구통계 정보, 평가자 간 합의 포함 |
All |
All |
|
7e |
성능 비교 시 기준(LLM, 인간, 기타 벤치마크/표준) 명시 |
All |
All |
|
주석(Annotation) |
8a |
주석을 작성한 경우, 텍스트 라벨링 방식, 구체적 가이드라인 및 예시 포함해 기술 |
All |
All |
|
8b |
주석자 수, 각 데이터 세트별 다중 주석 비율, 주석자 간 합의 등 포함 |
All |
All |
|
8c |
주석자 배경 및 경험, 라벨링에 관여한 모델 특성 등 정보 제공 |
All |
All |
|
프롬프트(Prompting) |
9a |
프롬프트 관련 연구일 경우, 프롬프트 설계, 선별, 선정과정 상세 기술 |
All |
All |
|
9b |
프롬프트 개발에 사용된 데이터 명시 |
All |
All |
|
요약(Summarization) |
10 |
요약 전 데이터 전처리 방법을 기술 |
All |
SS |
|
지시 조정/얼라인먼트(Instruction tuning/alignment) |
11 |
해당 전략 사용 시, 평가에 사용된 지침, 데이터, 인터페이스 및 평가 집단 특성 명시 |
M, D |
All |
|
연산 자원(Compute) |
12 |
연구 수행에 필요한 연산 자원 또는 그 대체지표(예: 사용한 기계 수, 소요시간, 비용, 추론시간, 초당 부동소수점 연산 횟수[FLOPS] 등) 보고 |
M, D, E |
All |
|
윤리 승인(Ethical approval) |
13 |
연구를 승인한 IRB 또는 윤리위원회 명칭, 참여자 동의 또는 동의 면제 여부 명시 |
All |
All |
|
오픈 사이언스(Open science) |
14a |
연구 자금 출처와 연구비 제공자의 역할 명시 |
All |
All |
|
14b |
모든 저자의 이해상충 및 재정 공개 선언 |
All |
All |
|
14c |
연구 프로토콜 공개 위치 명시 또는 미작성 명시 |
H |
All |
|
14d |
연구 등록정보(등록기관, 등록번호) 명시 또는 미등록 명시 |
H |
All |
|
14e |
연구 데이터 이용 가능성 상세 안내 |
All |
All |
|
14f |
연구결과 재현을 위한 코드 이용 가능성 상세 안내 |
All |
All |
|
공공 참여(Public involvement) |
15 |
설계, 수행, 보고, 해석, 결과 확산 등 과정에서 환자 및 공공 참여 내역 또는 미참여 사실 명시 |
H |
All |
|
결과(Results) |
|
|
|
|
|
참여자(Participants) |
16a |
환자/EHR 데이터 사용 시, 텍스트/EHR/환자 데이터의 흐름, 문서/질문/참여자 수(결과 보유ㆍ미보유 구분), 추적기간 등 기술 |
EH |
All |
|
16b |
환자/EHR 데이터 사용 시, 전체 및 각 데이터 출처/설정/개발ㆍ평가 분할별 특성, 주요 날짜, 주요 특성, 표본크기 등 보고 |
EH |
All |
|
16c |
임상결과 포함 LLM 평가 시, 개발 및 평가 데이터 간 주요 임상 변수 분포 비교(가능 시) |
EH |
All |
|
16d |
환자/EHR 데이터 사용 시, 각 분석(LLM 개발, 하이퍼파라미터 튜닝, 평가 등)별 참여자 및 결과 발생건수 명시 |
EH |
All |
|
성능(Performance) |
17 |
사전 지정한 평가지표(7a) 및/또는 인간 평가(7d)에 따라 LLM 성능 보고 |
All |
All |
|
LLM 업데이트(LLM updating) |
18 |
해당되는 경우, LLM 업데이트 결과와 이후 성능 보고 |
All |
All |
|
논의(Discussion) |
|
|
|
|
|
해석(Interpretation) |
19a |
주요 결과의 전반적 해석, 목표와 선행연구 맥락에서의 공정성 이슈 포함 |
All |
All |
|
한계(Limitations) |
19b |
연구 한계와 이에 따른 편향, 통계적 불확실성, 일반화 가능성에 미치는 영향 논의 |
All |
All |
|
맥락 내 LLM 활용성(Usability of the LLM in context) |
19c |
지정된 작업과 도메인 맥락에서 데이터를 사용하는 데 있어, 표현(representation), 결측(missingness) |
E, H |
All |
|
19d |
평가대상 적용사례의 의도된 사용 목적, 입력, 최종 사용자, 자율성/인간 감독 수준 정의 |
E, H |
All |
|
19e |
해당 시, LLM 적용 시 저품질 또는 이용 불가 입력 데이터의 평가ㆍ처리방법, 실제 임상에서의 LLM 활용성 기술 |
E, H |
All |
|
19f |
해당 시, 사용자가 입력 데이터 처리나 LLM 사용에 관여해야 하는 지와 필요한 전문성 수준 명시 |
E, H |
All |
|
19g |
향후 연구의 다음 단계, LLM의 적용 가능성 및 일반화 가능성 중심으로 논의 |
All |
All |
Table 3.
|
섹션(section) |
항목(item) |
체크리스트 항목(checklist item) |
연구설계(Research design) |
LLM 과업(LLM task) |
|
제목(Title) |
2a |
연구가 LLM의 개발, 미세조정, 성능 평가임을 밝히고, 해당 작업, 대상 집단, 예측하고자 하는 결과를 명시할 것 |
All |
All |
|
배경(Background) |
2b |
의료 맥락, 활용 사례, LLM 성능 개발 또는 평가의 근거를 간단히 설명할 것 |
E, H |
All |
|
목적(Objectives) |
2c |
연구목적을 명시하되, LLM 개발, 미세조정 및/또는 평가에 관한 것인지 포함할 것 |
All |
All |
|
2d |
연구환경의 주요 요소를 기술할 것 |
All |
All |
|
2e |
연구에 사용된 모든 데이터를 상세히 기술하고, 데이터 분할 및 선택적 사용 여부를 명확히 기술할 것 |
M, D, E |
All |
|
2f |
사용된 LLM의 이름과 버전을 명확히 밝힐 것 |
All |
All |
|
2g |
LLM 구축과정(미세조정, 보상 모델링, RLHF 등 포함)을 간단히 요약할 것 |
M, D |
All |
|
2h |
LLM이 수행한 구체적 작업(예: 의학 QA, 요약, 추출 등)을 기술하고, 최종 LLM의 주요 입력 및 출력을 강조할 것 |
All |
All |
|
방법(Methods) |
2i |
평가에 사용된 데이터 세트/집단과 평가 엔드포인트를 명시하고, 해당 정보를 학습/튜닝에서 배제했는지를 밝히며, LLM 성능 평가에 사용된 측정방법을 상세히 기술할 것 |
All |
All |
|
결과(Results) |
2j |
주요 결과의 전반적 보고와 해석을 제공할 것 |
All |
All |
|
논의(Discussion) |
2k |
결과에 비추어 발생할 수 있는 광범위한 시사점이나 우려 사항을 명확히 기술할 것 |
All |
All |
|
기타(Other) |
2l |
등록번호와 레지스트리/저장소 이름(해당 시)을 명시할 것 |
H |
All |
Box 1.
|
아래 정의와 설명은 TRIPOD-LLM의 특정 맥락 및 본 가이드라인에서의 용례에 한정된 것으로, 다른 연구 분야에는 그대로 적용되지 않을 수 있다. |
|
Attention mechanism(어텐션 메커니즘): 신경망에서 출력의 각 부분을 생성할 때 입력의 서로 다른 부분에 주의를 집중할 수 있도록 하여, 시퀀스 데이터 내 장거리 의존성 처리를 가능하게 하는 핵심 요소. |
|
Chain-of-thought prompting(사고 흐름 프롬프트): 모델이 복잡한 추론 과제를 단계별 사고과정으로 분해하여 처리하도록 유도하는 프롬프트 기법으로, 논리적ㆍ수리적 문제 해결력 향상에 도움을 준다. |
|
Confabulation(혼동 생성): Hallucination(환각)의 대체용어로, 의도하지 않게 허위정보를 생성하는 현상을 의미한다. |
|
Data leakage(데이터 누출): 모델학습 또는 미세조정 과정에서 테스트 데이터를 사용하는 것으로, 실제 성능보다 과대평가되는 결과를 초래한다. |
|
Decoder(디코더): 벡터화된 입력 데이터를 다시 텍스트 시퀀스로 변환하는 모델 구성요소. |
|
Autoregressive model(자기회귀모델): 시퀀스 내 앞선 요소를 바탕으로 다음 요소(예: 문장의 다음 단어)를 예측하는 트랜스포머 기반 모델. 최신 LLM (생성형 사전학습 변환기 등)은 대부분 자기회귀모델이다. |
|
Embedding(임베딩): 텍스트를 고차원 벡터 공간에 표현하여, 의미적으로 유사한 단어가 비슷한 벡터로 나타나게 하는 방식(‘벡터’ 참조). |
|
Encoder(인코더): 입력 데이터를 벡터화하거나 모델이 이해할 수 있는 표현으로 변환하는 모델의 구성요소. |
|
Encoder–decoder(인코더-디코더): 인코더와 디코더를 결합하여 입력 데이터를 출력으로 변환하는 모델 아키텍처 프레임워크. |
|
Few-shot learning(소수 예시 학습): 매우 적은 수의 예시만으로 모델이 작업을 효과적으로 수행할 수 있도록 하는 학습방법. 예시 수에 따라 one-shot learning 등으로 명명되기도 한다. |
|
Fine-tuning(미세조정): 사전 학습된 모델을 소규모 도메인 특화 데이터 세트로 추가 학습시켜 특정 작업에 특화하는 과정. |
|
GPT: 자연어 이해 및 생성을 위한 자기회귀 트랜스포머 기반 모델 계열. 문장 내 다음 단어 예측을 위한 사전 학습을 수행함. |
|
Hallucination(환각): 언어모델이 입력 데이터와 무관하거나 관련성이 적은 텍스트를 생성하는 현상. 허위, 부정확한 내용이 포함될 수 있다. |
|
In-context learning(문맥 내 학습): 추론 단계에서 프롬프트에 예시를 제공함으로써 모델이 새로운 작업을 학습하는 능력. |
|
Instruction tuning(지시 조정): 자연어 지시문과 바람직한 출력 쌍의 데이터 세트를 활용하여 모델이 다양한 지시문을 잘 따르도록 미세 조정하는 방법. |
|
Prompt(프롬프트): LLM에게 응답을 유도하기 위해 입력하는 질의 또는 지시문. |
|
Reinforcement learning(강화학습): LLM 개발에서 흔히 사용되는 머신러닝 기법으로, 행동에 대해 보상을 주어 모델이 인간 선호에 맞는 출력을 내도록 학습한다. |
|
Prompt engineering(프롬프트 엔지니어링): 원하는 출력을 얻기 위해 프롬프트를 설계ㆍ최적화하는 과정. 반복적 프롬프트, 예시 포함, 사고 흐름 프롬프트 등이 포함된다. |
|
Retrieval-augmented generation(검색 기반 생성): 외부 지식 베이스에서 정보를 검색해 생성과정에 결합, LLM이 최신 또는 도메인 특화 정보를 활용해 텍스트를 생성하도록 하는 방법. |
|
Temperature(온도 파라미터): Softmax 적용 전 로짓을 조정하여 예측의 무작위성을 제어하는 파라미터. 값이 높을수록 생성 텍스트의 다양성이 커진다. |
|
Tokenization(토크나이즈): 텍스트를 단어, 어절, 구 등 작은 단위(token)로 분할해 자연어 처리작업을 용이하게 하는 과정. |
|
Transformer(트랜스포머): NLP 분야의 혁신적 신경망 아키텍처. 자기 어텐션 메커니즘(self-attention)을 통해 시퀀스를 병렬 처리하고, 복잡한 종속관계도 효과적으로 포착한다. |
|
Vector(벡터): 데이터의 수치적 표현. LLM에서는 각 토큰(단어 조각)의 벡터가 주변 단어에 의해 영향을 받는 ‘문맥 임베딩(contextual embedding)’으로 나타난다. |
|
Zero-shot learning(제로샷 학습): 모델의 이해 및 일반화 능력에 기반해 명시적으로 학습한 적 없는 작업도 올바르게 수행하는 능력. |
Box 2.TRIPOD-LLM에서 TRIPOD-2015 및 TRIPOD+AI와 달라진 주요 내용 및 추가
|
LLM 보고를 위한 신규 체크리스트 도입: LLM의 고유한 특성과, 기존 AI 및 예측모델과 구별되는 특정 방법론을 반영하여 LLM 보고에 특화된 별도의 체크리스트가 개발되었다. |
|
생명력 있는(living) 가이드라인: 이 체크리스트는 생명력 있는 문서로 설계되어, 문헌 검토와 커뮤니티의 의견을 반영하여 정기적으로 업데이트된다. 이 방식은 본 분야의 빠른 발전속도를 반영하기 위한 것으로, 신속한 버전 관리, 사용자 테스트를 통한 개선, 적시 업데이트가 가능하도록 하였다. |
|
과업 특이적(task-specific) 지침 신설: 체크리스트에는 의료 분야의 다양한 LLM 응용에 따른 특정 도전과 필요를 해결하기 위한 과업별 지침이 새롭게 추가되었다. 이를 통해 연구 중인 LLM의 기능과 목적에 맞는 맞춤형의 관련성 높은 보고가 가능해진다. |
|
투명성과 공정성에 대한 강조 강화: 새로운 지침은 ‘투명성’과 ‘공정성’을 강조하며, 임상모델에 내재될 수 있는 사회적 편향의 인식 및 해결의 중요성을 부각하였다. 체크리스트는 이러한 개념을 전반에 통합하여, 모델 생애주기의 모든 단계에서 편향과 공정성이 고려되도록 하였다. |
|
모듈형(modular) 프레임워크: 새로운 지침은 모듈형 구조로, 각 연구에서 보고되는 연구설계 및 LLM 과업에 따라 요구사항이 달라진다. 이는 모델 개발부터 평가까지 생의학 LLM 연구의 다양한 응용과 접근법을 반영하여, 보다 특화된 보고 항목의 필요성에 대응하기 위함이다. |
References
- 1. Chen Z, Cano AH, Romanou A, Bonnet A, Matoba K, Salvi F, Pagliardini M, Fan S, Kopf A, Mohtashami A, Sallinen A. Meditron-70B: scaling medical pretraining for large language models. arXiv [Preprint] 2023 Nov 27. https://doi.org/10.48550/arXiv.2311.16079
- 2. OpenAI, Achiam J, Adler S, Agarwal S, Ahmad L, Akkaya I, Aleman FL, Almeida D, Altenschmidt J, Altman S, Anadkat S, Avila R, Babuschkin I, Balaji S, Balcom V, Baltescu P, Bao H, Bavarian M, Belgum J, Bello I, Berdine J, Bernadett-Shapiro G, Berner C, Bogdonoff L, Boiko O, Boyd M, Brakman AL, Brockman G, Brooks T, Brundage M, Button K, Cai T, Campbell R, Cann A, Carey B, Carlson C, Carmichael R, Chan B, Chang C, Chantzis F, Chen D, Chen S, Chen R, Chen J, Chen M, Chess B, Cho C, Chu C, Chung HW, Cummings D, Currier J, Dai Y, Decareaux C, Degry T, Deutsch N, Deville D, Dhar A, Dohan D, Dowling S, Dunning S, Ecoffet A, Eleti A, Eloundou T, Farhi D, Fedus L, Felix N, Fishman SP, Forte J, Fulford I, Gao L, Georges E, Gibson C, Goel V, Gogineni T, Goh G, Gontijo-Lopes R, Gordon J, Grafstein M, Gray S, Greene R, Gross J, Gu SS, Guo Y, Hallacy C, Han J, Harris J, He Y, Heaton M, Heidecke J, Hesse C, Hickey A, Hickey W, Hoeschele P, Houghton B, Hsu K, Hu S, Hu X, Huizinga J, Jain S, Jain S. GPT-4 technical report. arXiv [Preprint] 2023 Dec 19. https://doi.org/10.48550/arXiv.2303.08774
- 3. Singhal K, Azizi S, Tu T, Mahdavi SS, Wei J, Chung HW, Scales N, Tanwani A, Cole-Lewis H, Pfohl S, Payne P, Seneviratne M, Gamble P, Kelly C, Babiker A, Scharli N, Chowdhery A, Mansfield P, Demner-Fushman D, Aguera Y Arcas B, Webster D, Corrado GS, Matias Y, Chou K, Gottweis J, Tomasev N, Liu Y, Rajkomar A, Barral J, Semturs C, Karthikesalingam A, Natarajan V. Large language models encode clinical knowledge. Nature 2023;620:172-180. https://doi.org/10.1038/s41586-023-06291-2
- 4. Tai-Seale M, Baxter SL, Vaida F, Walker A, Sitapati AM, Osborne C, Diaz J, Desai N, Webb S, Polston G, Helsten T, Gross E, Thackaberry J, Mandvi A, Lillie D, Li S, Gin G, Achar S, Hofflich H, Sharp C, Millen M, Longhurst CA. AI-generated draft replies integrated into health records and physicians’ electronic communication. JAMA Netw Open 2024;7:e246565. https://doi.org/10.1001/jamanetworkopen.2024.6565
- 5. Tierney AA, Gayre G, Hoberman B, Mattern B, Ballesca M, Kipnis P, Liu V, Lee K. Ambient artificial intelligence scribes to alleviate the burden of clinical documentation. NEJM Catal Innov Care Deliv 2024;5:CAT.23.0404. https://doi.org/10.1056/CAT.23.0404
- 6. Jiang LY, Liu XC, Nejatian NP, Nasir-Moin M, Wang D, Abidin A, Eaton K, Riina HA, Laufer I, Punjabi P, Miceli M, Kim NC, Orillac C, Schnurman Z, Livia C, Weiss H, Kurland D, Neifert S, Dastagirzada Y, Kondziolka D, Cheung AT, Yang G, Cao M, Flores M, Costa AB, Aphinyanaphongs Y, Cho K, Oermann EK. Health system-scale language models are all-purpose prediction engines. Nature 2023;619:357-362. https://doi.org/10.1038/s41586-023-06160-y
- 7. Cohen MK, Kolt N, Bengio Y, Hadfield GK, Russell S. Regulating advanced artificial agents. Science 2024;384:36-38. https://doi.org/10.1126/science.adl0625
- 8. Mesko B, Topol EJ. The imperative for regulatory oversight of large language models (or generative AI) in healthcare. NPJ Digit Med 2023;6:120. https://doi.org/10.1038/s41746-023-00873-0
- 9. Collins GS, Reitsma JB, Altman DG, Moons KG. Transparent reporting of a multivariable prediction model for individual prognosis or diagnosis (TRIPOD): the TRIPOD statement. BMJ 2015;350:g7594. https://doi.org/10.1136/bmj.g7594
- 10. EQUATOR Network. Reporting guidelines [Internet]. EQUATOR Network [cited 2024 Jun 1]. Available from: https://www.equator-network.org/reporting-guidelines/
- 11. Collins GS, Moons KG, Dhiman P, Riley RD, Beam AL, Van Calster B, Ghassemi M, Liu X, Reitsma JB, van Smeden M, Boulesteix AL, Camaradou JC, Celi LA, Denaxas S, Denniston AK, Glocker B, Golub RM, Harvey H, Heinze G, Hoffman MM, Kengne AP, Lam E, Lee N, Loder EW, Maier-Hein L, Mateen BA, McCradden MD, Oakden-Rayner L, Ordish J, Parnell R, Rose S, Singh K, Wynants L, Logullo P. TRIPOD+AI statement: updated guidance for reporting clinical prediction models that use regression or machine learning methods. BMJ 2024;385:e078378. https://doi.org/10.1136/bmj-2023-078378
- 12. Liu X, Cruz Rivera S, Moher D, Calvert MJ, Denniston AK. Reporting guidelines for clinical trial reports for interventions involving artificial intelligence: the CONSORT-AI extension. Nat Med 2020;26:1364-1374. https://doi.org/10.1038/s41591-020-1034-x
- 13. Vasey B, Nagendran M, Campbell B, Clifton DA, Collins GS, Denaxas S, Denniston AK, Faes L, Geerts B, Ibrahim M, Liu X, Mateen BA, Mathur P, McCradden MD, Morgan L, Ordish J, Rogers C, Saria S, Ting DS, Watkinson P, Weber W, Wheatstone P, McCulloch P. Reporting guideline for the early-stage clinical evaluation of decision support systems driven by artificial intelligence: DECIDE-AI. Nat Med 2022;28:924-933. https://doi.org/10.1038/s41591-022-01772-9
- 14. Norgeot B, Quer G, Beaulieu-Jones BK, Torkamani A, Dias R, Gianfrancesco M, Arnaout R, Kohane IS, Saria S, Topol E, Obermeyer Z, Yu B, Butte AJ. Minimum information about clinical artificial intelligence modeling: the MI-CLAIM checklist. Nat Med 2020;26:1320-1324. https://doi.org/10.1038/s41591-020-1041-y
- 15. Agrawal M, Hegselmann S, Lang H, Kim Y, Sontag D. Large language models are few-shot clinical information extractors. In: Goldberg Y, Kozareva Z, Zhang Y, editors. Proceedings of the 2022 Conference on Empirical Methods in Natural Language Processing; 2022 Dec; Abu Dhabi, United Arab Emirates. Association for Computational Linguistics; 2022. p. 1998-2022. https://doi.org/10.18653/v1/2022.emnlp-main.130
- 16. Liu X, McDuff D, Kovacs G, Galatzer-Levy I, Sunshine J, Zhan J, Poh MZ, Liao S, Di Achille P, Patel S. Large language models are few-shot health learners. arXiv [Preprint] 2023 May 24. https://doi.org/10.48550/arXiv.2305.15525
- 17. Salazar J, Liang D, Nguyen TQ, Kirchhoff K. Masked language model scoring. In: Jurafsky D, Chai J, Schluter N, Tetreault J, editors. Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics; 2020 Jul; Online. Association for Computational Linguistics; 2022. p. 2699-2712. https://doi.org/10.18653/v1/2020.acl-main.240
- 18. Wang A, Singh A, Michael J, Hill F, Levy O, Bowman S. GLUE: a multi-task benchmark and analysis platform for natural language understanding. In: Linzen T, Chrupala G, Alishahi A, editors. Proceedings of the 2018 EMNLP Workshop BlackboxNLP: Analyzing and Interpreting Neural Networks for NLP; 2018 Nov; Brussels, Belgium. Association for Computational Linguistics; 2018. p. 353-355. https://doi.org/10.18653/v1/W18-5446
- 19. Papineni K, Roukos S, Ward T, Zhu WJ. Bleu: a method for automatic evaluation of machine translation. In: Isabelle P, Charniak E, Lin D, editors. Proceedings of the 40th Annual Meeting of the Association for Computational Linguistics; 2002 Jul; Philadelphia, USA. Association for Computational Linguistics; 2002. p. 311-318. https://doi.org/10.3115/1073083.1073135
- 20. Zack T, Lehman E, Suzgun M, Rodriguez JA, Celi LA, Gichoya J, Jurafsky D, Szolovits P, Bates DW, Abdulnour RE, Butte AJ, Alsentzer E. Assessing the potential of GPT-4 to perpetuate racial and gender biases in health care: a model evaluation study. Lancet Digit Health 2024;6:e12-e22. https://doi.org/10.1016/S2589-7500(23)00225-X
- 21. Chen S, Guevara M, Moningi S, Hoebers F, Elhalawani H, Kann BH, Chipidza FE, Leeman J, Aerts HJWL, Miller T, Savova GK, Gallifant J, Celi LA, Mak RH, Lustberg M, Afshar M, Bitterman DS. The effect of using a large language model to respond to patient messages. Lancet Digit Health 2024;6:e379-e381. https://doi.org/10.1016/S2589-7500(24)00060-8
- 22. Peng C, Yang X, Chen A, Smith KE, PourNejatian N, Costa AB, Martin C, Flores MG, Zhang Y, Magoc T, Lipori G, Mitchell DA, Ospina NS, Ahmed MM, Hogan WR, Shenkman EA, Guo Y, Bian J, Wu Y. A study of generative large language model for medical research and healthcare. NPJ Digit Med 2023;6:210. https://doi.org/10.1038/s41746-023-00958-w
- 23. Zakka C, Shad R, Chaurasia A, Dalal AR, Kim JL, Moor M, Fong R, Phillips C, Alexander K, Ashley E, Boyd J, Boyd K, Hirsch K, Langlotz C, Lee R, Melia J, Nelson J, Sallam K, Tullis S, Vogelsong MA, Cunningham JP, Hiesinger W. Almanac: retrieval-augmented language models for clinical medicine. NEJM AI 2024;1:10.1056/aioa2300068. https://doi.org/10.1056/aioa2300068
- 24. Keloth VK, Hu Y, Xie Q, Peng X, Wang Y, Zheng A, Selek M, Raja K, Wei CH, Jin Q, Lu Z, Chen Q, Xu H. Advancing entity recognition in biomedicine via instruction tuning of large language models. Bioinformatics 2024;40:btae163. https://doi.org/10.1093/bioinformatics/btae163
- 25. Guevara M, Chen S, Thomas S, Chaunzwa TL, Franco I, Kann BH, Moningi S, Qian JM, Goldstein M, Harper S, Aerts HJ, Catalano PJ, Savova GK, Mak RH, Bitterman DS. Large language models to identify social determinants of health in electronic health records. NPJ Digit Med 2024;7:6. https://doi.org/10.1038/s41746-023-00970-0
- 26. Jin Q, Yang Y, Chen Q, Lu Z. GeneGPT: augmenting large language models with domain tools for improved access to biomedical information. Bioinformatics 2024;40:btae075. https://doi.org/10.1093/bioinformatics/btae075
- 27. Goh E, Gallo R, Hom J, Strong E, Weng Y, Kerman H, Cool JA, Kanjee Z, Parsons AS, Ahuja N, Horvitz E, Yang D, Milstein A, Olson AP, Rodman A, Chen JH. Large language model influence on diagnostic reasoning: a randomized clinical trial. JAMA Netw Open 2024;7:e2440969. https://doi.org/10.1001/jamanetworkopen.2024.40969
- 28. Zaretsky J, Kim JM, Baskharoun S, Zhao Y, Austrian J, Aphinyanaphongs Y, Gupta R, Blecker SB, Feldman J. Generative artificial intelligence to transform inpatient discharge summaries to patient-friendly language and format. JAMA Netw Open 2024;7:e240357. https://doi.org/10.1001/jamanetworkopen.2024.0357
- 29. Han L, Erofeev G, Sorokina I, Gladkoff S, Nenadic G. Examining large pre-trained language models for machine translation: what you don’t know about it. Proceedings of the Seventh Conference on Machine Translation (WMT); 2022 Dec; Abu Dhabi, United Arab Emirates (Hybrid). Association for Computational Linguistics; 2022. p. 908-919.
- 30. Yoon W, Chen S, Gao Y, Zhao Z, Dligach D, Bitterman DS, Afshar M, Miller T. LCD benchmark: long clinical document benchmark on mortality prediction for language models. J Am Med Inform Assoc 2025;32:285-295. https://doi.org/10.1093/jamia/ocae287
- 31. Goodman KE, Yi PH, Morgan DJ. AI-generated clinical summaries require more than accuracy. JAMA 2024;331:637-638. https://doi.org/10.1001/jama.2024.0555
- 32. Gallifant J, Fiske A, Levites Strekalova YA, Osorio-Valencia JS, Parke R, Mwavu R, Martinez N, Gichoya JW, Ghassemi M, Demner-Fushman D, McCoy LG, Celi LA, Pierce R. Peer review of GPT-4 technical report and systems card. PLOS Digit Health 2024;3:e0000417. https://doi.org/10.1371/journal.pdig.0000417
- 33. Wornow M, Xu Y, Thapa R, Patel B, Steinberg E, Fleming S, Pfeffer MA, Fries J, Shah NH. The shaky foundations of large language models and foundation models for electronic health records. NPJ Digit Med 2023;6:135. https://doi.org/10.1038/s41746-023-00879-8
- 34. Chang CT, Farah H, Gui H, Rezaei SJ, Bou-Khalil C, Park YJ, Swaminathan A, Omiye JA, Kolluri A, Chaurasia A, Lozano A, Heiman A, Jia AS, Kaushal A, Jia A, Iacovelli A, Yang A, Salles A, Singhal A, Narasimhan B, Belai B, Jacobson BH, Li B, Poe CH, Sanghera C, Zheng C, Messer C, Kettud DV, Pandya D, Kaur D, Hla D, Dindoust D, Moehrle D, Ross D, Chou E, Lin E, Haredasht FN, Cheng G, Gao I, Chang J, Silberg J, Fries JA, Xu J, Jamison J, Tamaresis JS, Chen JH, Lazaro J, Banda JM, Lee JJ, Matthys KE, Steffner KR, Tian L, Pegolotti L, Srinivasan M, Manimaran M, Schwede M, Zhang M, Nguyen M, Fathzadeh M, Zhao Q, Bajra R, Khurana R, Azam R, Bartlett R, Truong ST, Fleming SL, Raj S, Behr S, Onyeka S, Muppidi S, Bandali T, Eulalio TY, Chen W, Zhou X, Ding Y, Cui Y, Tan Y, Liu Y, Shah NH, Daneshjou R. Red teaming large language models in medicine: real-world insights on model behavior. medRxiv [Preprint] 2024 Apr 7. https://doi.org/10.1101/2024.04.05.24305411
- 35. Gallifant J, Chen S, Moreira PJ, Munch N, Gao M, Pond J, Celi LA, Aerts H, Hartvigsen T, Bitterman D. Language models are surprisingly fragile to drug names in biomedical benchmarks. In: Al-Onaizan Y, Bansal M, Chen YN, editors. Proceedings of the Findings of the Association for Computational Linguistics: EMNLP 2024; 2024 Nov; Miami, USA. Association for Computational Linguistics; 2024. p. 12448-12465. https://doi.org/10.18653/v1/2024.findings-emnlp.726
- 36. Boyd E. Microsoft and Epic expand AI collaboration to accelerate generative AI’s impact in healthcare, addressing the industry’s most pressing needs [Internet]. Microsoft; 2023 [cited 2024 Jun 1]. Available from: https://blogs.microsoft.com/blog/2023/08/22/microsoft-and-epic-expand-ai-collaboration-to-accelerate-generative-ais-impact-in-healthcare-addressing-the-industrys-most-pressing-needs/
- 37. Moreno AC, Bitterman DS. Toward Clinical-Grade Evaluation of Large Language Models. Int J Radiat Oncol Biol Phys 2024;118:916-920. https://doi.org/10.1016/j.ijrobp.2023.11.012
- 38. Welch Medical Library. Evidence based medicine: evidence grading & reporting [Internet]. Johns Hopkins University [cited 2024 Jun 1]. Available from: https://browse.welch.jhmi.edu/EBM/EBM_EvidenceGrading
- 39. Guyatt GH, Oxman AD, Kunz R, Vist GE, Falck-Ytter Y, Schunemann HJ; GRADE Working Group. What is “quality of evidence” and why is it important to clinicians? BMJ 2008;336:995-998. https://doi.org/10.1136/bmj.39490.551019.BE
- 40. Huo B, Cacciamani GE, Collins GS, McKechnie T, Lee Y, Guyatt G. Reporting standards for the use of large language model-linked chatbots for health advice. Nat Med 2023;29:2988. https://doi.org/10.1038/s41591-023-02656-2
- 41. Cacciamani GE, Collins GS, Gill IS. ChatGPT: standard reporting guidelines for responsible use. Nature 2023;618:238. https://doi.org/10.1038/d41586-023-01853-w
- 42. El Mikati IK, Khabsa J, Harb T, Khamis M, Agarwal A, Pardo-Hernandez H, Farran S, Khamis AM, El Zein O, El-Khoury R, Schünemann HJ, Akl EA, Alonso-Coello P, Alper BS, Amer YS, Arayssi T, Barker JM, Bouakl I, Boutron I, Brignardello-Petersen R, Carandang K, Chang S, Chen Y, Cuker A, El-Jardali F, Florez I, Ford N, Grove J, Guyatt GH, Hazlewood GS, Kredo T, Lamontagne F, Langendam MW, Lewin S, Macdonald H, McFarlane E, Meerpohl J, Munn Z, Murad MH, Mustafa RA, Neumann I, Nieuwlaat R, Nowak A, Pardo JP, Qaseem A, Rada G, Righini M, Rochwerg B, Rojas-Reyes MX, Siegal D, Siemieniuk R, Singh JA, Skoetz N, Sultan S, Synnot A, Tugwell P, Turner A, Turner T, Venkatachalam S, Welch V, Wiercioch W. A framework for the development of living practice guidelines in health care. Ann Intern Med 2022;175:1154-1160. https://doi.org/10.7326/M22-0514
- 43. Cochrane Community. Living systematic reviews [Internet]. Cochrane [cited 2024 Jun 1]. Available from: https://community.cochrane.org/review-development/resources/living-systematic-reviews
- 44. Akl EA, Meerpohl JJ, Elliott J, Kahale LA, Schünemann HJ. Living systematic reviews: 4. Living guideline recommendations. J Clin Epidemiol 2017;91:47-53. https://doi.org/10.1016/j.jclinepi.2017.08.009
- 45. Fraile Navarro D, Cheyne S, Hill K, McFarlane E, Morgan RL, Murad MH, Mustafa RA, Sultan S, Tunnicliffe DJ, Vogel JP, White H, Turner T. Methods for living guidelines: early guidance based on practical experience. Paper 5: decisions on methods for evidence synthesis and recommendation development for living guidelines. J Clin Epidemiol 2023;155:118-128. https://doi.org/10.1016/j.jclinepi.2022.12.022
- 46. Bedoya AD, Economou-Zavlanos NJ, Goldstein BA, Young A, Jelovsek JE, O’Brien C, Parrish AB, Elengold S, Lytle K, Balu S, Huang E, Poon EG, Pencina MJ. A framework for the oversight and local deployment of safe and high-quality prediction models. J Am Med Inform Assoc 2022;29:1631-1636. https://doi.org/10.1093/jamia/ocac078
- 47. Omiye JA, Gui H, Rezaei SJ, Zou J, Daneshjou R. Large language models in medicine: the potentials and pitfalls : a narrative review. Ann Intern Med 2024;177:210-220. https://doi.org/10.7326/M23-2772
- 48. Chen S, Gallifant J, Gao M, Moreira P, Munch N, Muthukkumar A, Rajan A, Kolluri J, Fiske A, Hastings J, Aerts H, Anthony B, Celi LA, La Cava WG, Bitterman DS. Cross-care: assessing the healthcare implications of pre-training data on language model bias. Adv Neural Inf Process Syst 2024;37:23756-23795.
- 49. Hansen LH, Andersen N, Gallifant J, McCoy LG, Stone JK, Izath N, Aguirre-Jerez M, Bitterman DS, Gichoya J, Celi LA. Seeds of stereotypes: a large-scale textual analysis of race and gender associations with diseases in online sources. arXiv [Preprint] 2024 May 8. https://doi.org/10.48550/arXiv.2405.05049
- 50. Biderman S, Schoelkopf H, Anthony QG, Bradley H, O’Brien K, Hallahan E, Khan MA, Purohit S, Prashanth US, Raff E, Skowron A, Sutawika L, Van Der Wal O. Pythia: a suite for analyzing large language models across training and scaling. Proceedings of the 40th International Conference on Machine Learning; 2023 Jul 23-29; Honolulu, USA. PMLR; 2023.
- 51. Bowman SR, Hyun J, Perez E, Chen E, Pettit C, Heiner S, Lukosiute K, Askell A, Jones A, Chen A, Goldie A, Mirhoseini A, McKinnon C, Olah C, Amodei D, Amodei D, Drain D, Li D, Tran-Johnson E, Kernion J, Kerr J, Mueller J, Ladish J, Landau J, Ndousse K, Lovitt L, Elhage N, Schiefer N, Joseph N, Mercado N, DasSarma N, Larson R, McCandlish S, Kundu S, Scott Johnston, Kravec S, El Showk S, Fort S, Telleen-Lawton T, Brown T, Henighan T, Hume T, Bai Y, Hatfield-Dodds Z, Mann B, Kaplan J. Measuring progress on scalable oversight for large language models. arXiv [Preprint] 2022 Nov 11. https://doi.org/10.48550/arXiv.2211.03540
- 52. McAleese N, Pokorny RM, Uribe JF, Nitishinskaya E, Trebacz M, Leike J. LLM critics help catch LLM bugs. arXiv [Preprint] 2024 Jun 28. https://doi.org/10.48550/arXiv.2407.00215
- 53. Burns C, Izmailov P, Kirchner JH, Baker B, Gao L, Aschenbrenner L, Chen Y, Ecoffet A, Joglekar M, Leike J, Sutskever I. Weak-to-strong generalization: eliciting strong capabilities with weak supervision. arXiv [Preprint] 2023 Dec 14. https://doi.org/10.48550/arXiv.2312.09390
- 54. Chen S, Li Y, Lu S, Van H, Aerts HJ, Savova GK, Bitterman DS. Evaluating the ChatGPT family of models for biomedical reasoning and classification. J Am Med Inform Assoc 2024;31:940-948. https://doi.org/10.1093/jamia/ocad256
- 55. Chen S, Kann BH, Foote MB, Aerts HJ, Savova GK, Mak RH, Bitterman DS. Use of artificial intelligence chatbots for cancer treatment information. JAMA Oncol 2023;9:1459-1462. https://doi.org/10.1001/jamaoncol.2023.2954
- 56. Lu MY, Chen B, Williamson DF, Chen RJ, Liang I, Ding T, Jaume G, Odintsov I, Le LP, Gerber G, Parwani AV, Zhang A, Mahmood F. A visual-language foundation model for computational pathology. Nat Med 2024;30:863-874. https://doi.org/10.1038/s41591-024-02856-4
- 57. Kocak B, Akinci D’Antonoli T, Mercaldo N, Alberich-Bayarri A, Baessler B, Ambrosini I, Andreychenko AE, Bakas S, Beets-Tan RG, Bressem K, Buvat I, Cannella R, Cappellini LA, Cavallo AU, Chepelev LL, Chu LC, Demircioglu A, deSouza NM, Dietzel M, Fanni SC, Fedorov A, Fournier LS, Giannini V, Girometti R, Groot Lipman KB, Kalarakis G, Kelly BS, Klontzas ME, Koh DM, Kotter E, Lee HY, Maas M, Marti-Bonmati L, Muller H, Obuchowski N, Orlhac F, Papanikolaou N, Petrash E, Pfaehler E, Pinto Dos Santos D, Ponsiglione A, Sabater S, Sardanelli F, Seebock P, Sijtsema NM, Stanzione A, Traverso A, Ugga L, Vallieres M, van Dijk LV, van Griethuysen JJ, van Hamersvelt RW, van Ooijen P, Vernuccio F, Wang A, Williams S, Witowski J, Zhang Z, Zwanenburg A, Cuocolo R. METhodological RadiomICs Score (METRICS): a quality scoring tool for radiomics research endorsed by EuSoMII. Insights Imaging 2024;15:8. https://doi.org/10.1186/s13244-023-01572-w
- 58. Lambin P, Leijenaar RT, Deist TM, Peerlings J, de Jong EE, van Timmeren J, Sanduleanu S, Larue RT, Even AJ, Jochems A, van Wijk Y, Woodruff H, van Soest J, Lustberg T, Roelofs E, van Elmpt W, Dekker A, Mottaghy FM, Wildberger JE, Walsh S. Radiomics: the bridge between medical imaging and personalized medicine. Nat Rev Clin Oncol 2017;14:749-762. https://doi.org/10.1038/nrclinonc.2017.141
- 59. Shah NH, Halamka JD, Saria S, Pencina M, Tazbaz T, Tripathi M, Callahan A, Hildahl H, Anderson B. A nationwide network of health ai assurance laboratories. JAMA 2024;331:245-249. https://doi.org/10.1001/jama.2023.26930
- 60. Diaz N. Epic releases AI validation suite [Internet]. Becker’s Hospital Review; 2024 [cited 2024 May 23]. Available from: https://www.beckershospitalreview.com/ehrs/epic-releases-ai-validation-suite.html
- 61. Epic-open-source/seismometer [Internet]. GitHub; 2024 [cited 2024 May 23]. Available from: https://github.com/epic-open-source/seismometer
- 62. National Institute of Standards and Technology (NIST). U.S. Artificial Intelligence Safety Institute [Internet]. NIST; 2023 [cited 2024 May 23]. Available from: https://www.nist.gov/aisi
- 63. Federal Register. Health data, technology, and interoperability: certification program updates, algorithm transparency, and information sharing [Internet]. Federal Register; 2024 [cited 2024 May 23]. Available from: https://www.federalregister.gov/documents/2024/01/09/2023-28857/health-data-technology-and-interoperability-certification-program-updates-algorithm-transparency-and
- 64. EU Artificial Intelligence Act. The AI Act Explorer [Internet]. EU Artificial Intelligence Act; 2024 [cited 2024 May 23]. Available from: [cited 2024 May 23]. Available from: https://artificialintelligenceact.eu/ai-act-explorer/
 , Majid Afshar4,29
, Majid Afshar4,29 , Saleem Ameen1,5,6,29
, Saleem Ameen1,5,6,29 , Yindalon Aphinyanaphongs7,29
, Yindalon Aphinyanaphongs7,29 , Shan Chen3,8,29
, Shan Chen3,8,29 , Giovanni Cacciamani9,10,29
, Giovanni Cacciamani9,10,29 , Dina Demner-Fushman11,29, Dmitriy Dligach12,29
, Dina Demner-Fushman11,29, Dmitriy Dligach12,29 , Roxana Daneshjou13,14,29
, Roxana Daneshjou13,14,29 , Chrystinne Fernandes1,29
, Chrystinne Fernandes1,29 , Lasse Hyldig Hansen1,15,29
, Lasse Hyldig Hansen1,15,29 , Adam Landman16,29
, Adam Landman16,29 , Lisa Lehmann16,29, Liam G. McCoy17,29, Timothy Miller18,29
, Lisa Lehmann16,29, Liam G. McCoy17,29, Timothy Miller18,29 , Amy Moreno19,29
, Amy Moreno19,29 , Nikolaj Munch1,15,29, David Restrepo1,20,29
, Nikolaj Munch1,15,29, David Restrepo1,20,29 , Guergana Savova18,29
, Guergana Savova18,29 , Renato Umeton21,29
, Renato Umeton21,29 , Judy Wawira Gichoya22,29
, Judy Wawira Gichoya22,29 , Gary S. Collins23,24
, Gary S. Collins23,24 , Karel G. M. Moons25,26
, Karel G. M. Moons25,26 , Leo A. Celi1,27,28
, Leo A. Celi1,27,28 , Danielle S. Bitterman3,8,*
, Danielle S. Bitterman3,8,*