Abstract
멘델 무작위화(Mendelian randomization, MR) 연구는 조절 가능한 노출(modifiable exposure)이 건강결과에 미치는 인과효과(causal effect)를 더 잘 이해하게 해 주지만, 그 근거는 종종 보고가 불충분함으로 인하여연구 결과의 해석과 적용에 한계가 있을 수 있다. 보고지침은 흔히 무슨 연구를 하고 무엇을 발견했는지 독자가 쉽게 이해하도록 돕는다. STROBE-MR(관찰연구의 멘델 무작위화를 활용한 보고지침)은 MR 연구를 명확하고 투명하게 보고하도록 돕는다. STROBE-MR을 논문 작성에 활용하면 독자, 심사자, 학술지 편집인이 MR 연구의 보고 품질과 완성도를 평가하는 데 도움이 될 것이다. 이 글은 STROBE-MR 체크리스트 20개 항목의 의미와 근거를 설명하고, 각 항목마다 사례를 제시해 독자가 잘 이해할 수 있는 논문 작성법을 설명하려고 하였다.
요약
관찰연구에서 멘델 무작위화(Mendelian randomization, MR) 연구는 편향(bias)의 위험을 줄이면서 노출과 결과 간 인과적 관련성을 알 수 있게 한다. MR 연구 보고에 관한 합의가 부족하고, 보고의 질도 일관되지 않았다. 다수의 MR 논문은 MR의 각 가정을 명시하거나 점검하지 않았고, 자료 출처에 대한 세부 정보도 불충분하게 보고하였다. STROBE-MR(관찰연구의 멘델 무작위화를 활용한 보고지침)은 MR 연구를 쉽게 이해하도록 20개 보고 항목으로 구성된 체크리스트를 제시하였다. 이 논문은 각 체크리스트 항목의 근거를 설명하고, 명확한 보고 예를 들었다. MR 연구의 저자, 심사자, 학술지 편집인은 STROBE-MR을 활용하여 MR 연구 보고의 질을 높이기를 권한다.
서론
관찰연구는 흔히 노출과 건강결과 간의 관련성을 다룬다. 그러나 역학 연구에서 보고되는 이러한 관련성은 인과적 추론을 하기에는 충분하지 않은 경우가 많으며, 결과와 노출 모두에 영향을 미치는 제3의 요인에 의한 교란 또는 다른 형태의 편향으로 생성될 수 있다[
1-
3]. 예를 들어, 알코올 섭취는 흡연, 권장하지 않는 식습관, 운동 부족 등 많은 잠재적 교란요인과 관련될 수 있다. 반대로 건강이 좋지 않으면 알코올 섭취의 감소나 중단이 뒤따를 수 있어, 알코올 섭취가 이후 건강에 미치는 영향을 연구할 때 역인과(reverse causality)로 인한 편향 가능성이 생긴다[
4,
5]. 이러한 편향을 완화하려는 여러 접근법이 개발되어 왔다[
6]. 예컨대 도구변수 분석(instrumental variable methods)은 노출과 관련되어 있으나, 노출을 통하지 않고는 결과에 영향을 미치지 않는다[
6,
7].
지난 10여 년간의 유전체 분석 기술의 발전은 유전변이와 관련 노출, 형질, 건강결과 사이의 수천 개 재현 가능한 관련성을 확인하게 하였다. 이러한 유전변이는 멘델 무작위화(Mendelian randomization, MR)라 불리는 연구방법을 통해, 조절 가능한 노출이 질병에 미치는 영향을 분석하는 도구변수로 사용할 수 있다[
8]. MR 연구는 조절 가능한 노출과 깊이 관련된 유전변이를 이용해, 노출이 다양한 건강ㆍ사회ㆍ경제적 결과에 미치는 영향을 이해하도록 한다. 유전변이는 부모로부터 무작위로 자식에게 유전되므로, 노출–결과 관련성을 교란할 수 있는 많은 요인이 유전변이에 영향을 미치지 못한다. 유전변이는 일반적으로 결과에 의해 영향을 받지 않으므로 역인과의 영향에서도 비교적 자유롭다. 따라서 MR은 교란과 역인과에서 비롯될 수 있는 잠재적 편향을 줄이면서 노출과 결과의 관련성을 연구할 기회를 제공한다[
9].
이러한 특성은 유전변이를 도구변수의 적절한 후보로 만들며, 조절 가능한 노출이 결과에 미치는 인과효과 추정에 기여한다[
7]. 예를 들어, 알코올 탈수소효소 1B 유전자(ADH1B)의 rs1229984 변이는 심혈관질환에서 알코올의 인과적 역할을 조사하는 도구로 사용되어 왔다[
10]. 이러한 장점에 힘입어 MR 연구는 빠르게 확산되었으며, 질병 원인을 보다 정확히 이해하고 예방, 치료 저략 수립에 기여하기 시작하였다. MR은 유전변이를 사용해 도구변수 추정을 생성하는 연구(Box 1,
Table 1)로만 국한되지 않지만, 현재 문헌에서는 이러한 유형의 연구가 대다수를 이룬다. MR에서 흔히 사용되는 용어의 용어집은
Table 2에 제시되어 있으며, 추가 용어와 설명은 오픈 액세스 MR 사전에 수록되어 있다[
21].
Box 1. 멘델 무작위화(MR)의 범위와 STROBE-MR 체크리스트
MR은 일반적으로 유전 변이를 도구변수로 사용하지만, 그러한 연구에만 한정되지 않는다. 실제로 “멘델 무작위화”라는 용어는 소아 악성종양 치료에서의 골수이식 연구를 위해 1991년에 도입되었다[
11,
12]. 기본 개념은 human leukocyte antigen (HLA)가 일치하는 형제자매가 있는 아동은 그렇지 않은 아동보다 골수이식을 받을 가능성이 높다는 점이었다. 형제자매의 유무(이상적으로는 형제자매 수까지 고려하여)에 따라 결과를 분석하는 것은 무작위배정 임상시험에서 치료 의도(intention-to-treat) 분석과 유사하다[
11,
12]. HLA 일치 형제자매를 “우연히” 가질 수 있다는 사실은 골수이식의 유전적 도구로도 기능할 수 있으므로, 이식이 암 결과에 미치는 효과를 추론하는 데 활용될 수 있다. 이러한 접근은 이후에도 계속 사용되어 왔다[
13-
15]. 초기에는 MR을 “질병 위험 또는 기타 결과에 영향을 미치는 조절 가능한 노출의 인과추론을 강화하기 위해 생식계열(germline) 유전변이를 사용하는 것”으로 정의하였다[
16]. 이러한 광의의 정의에는 예를 들어 유전자-공변량 상호작용 연구(종종 환경을 공변량으로 함)가 포함되며, 여기서 상호작용은 관심 노출에 대한 도구변수로 간주될 수 없다[
17,
18]. 또한 쌍생아 연구와 같이 멘델 유전의 기본 원리를 사용하는 다른 연구설계도 MR의 한 형태로 간주할 수 있다. 이러한 사례 중 하나는 (평균적으로) 태아기 테스토스테론 수치가 더 높음을 나타내는 지표로 남자 쌍생아를 대상으로 하여 테스토스테론이 신경발달적 특성에 미치는 영향을 평가하였다[
19]. MR 연구는 단일염기다형성과 결과 간 관련성을 단순히 시험해 특정 노출이 질병에 영향을 미치는지에 대한 근거를 제공하는 수준부터, 도구변수 분석으로부터 특정 효과 추정치를 산출하는 수준까지 폭넓다.
STROBE-MR 현재 도구변수 체계 내에서 수행되는 대다수 MR 연구를 주된 대상으로 한다. 노출에 대한 도구를 사용하지 않는 MR 연구(예: 유전자-환경 상호작용 연구)나, 유전변이를 도구변수 체계에서 사용하지만 도구변수 추정치를 보고하지 않는 MR 연구(예: 이식에서의 형제자매 적합성 연구)의 경우, STROBE-MR 항목 중 일부는 적용되지 않을 수 있다. 그렇지만 체크리스트는 여전히 유용한 지침을 제공한다.
Table 1은 STROBE-MR이 다루는 연구설계와 다루지 않는 연구설계 설명이다.
MR 연구 보고의 강화
MR 적용과 방법이 성장하고 MR 결과의 중요성이 커졌음에도, MR 연구 보고방식에 대한 합의는 부족하다. 그 결과, MR 연구의 보고 질은 일관되지 않았다. 실증적 근거에 따르면[
22-
24], 많은 MR 연구 보고가 MR 방법의 다양한 가정을 명확히 기술하거나 점검하지 않았고, 자료 출처에 대한 세부 정보를 불충분하게 보고하여 결과의 질과 신뢰성을 평가하기 어렵게 만든다.
관찰연구를 위한 STROBE (strengthening the reporting of observational studies in epidemiology) 지침은 역학의 세 가지 주요 연구설계(코호트, 환자-대조군, 단면연구)를 대상으로 개발되었다[
25,
26]. 그러나 STROBE의 일부 항목은 지나치게 일반적이거나 MR 연구에 적용되지 않으며, MR 연구에 중요한 항목이 누락되어 있기도 하다. MR 연구 보고를 개선하기 위해, 우리는 STROBE 지침에 기반을 두되 MR 연구설계에 초점을 맞춘 별도의 체크리스트를 개발하였고, 그 결과가 STROBE-MR 보고지침(strengthening the reporting of observational studies in epidemiology using Mendelian randomization)이다[
27] (
Table 3). STROBE 체크리스트와 유사하게, STROBE-MR 항목은 논문의 제목, 초록, 서론, 방법, 결과, 고찰에 해당한다.
STROBE-MR의 개발, 범위, 활용
자세한 내용은 다른 곳에 기술되어 있으며[
27], 우리는 의학 연구 보고지침 개발을 위한 권고를 따라[
28], 2018년에 작업을 시작하였다. MR 방법론 전문가, 기존 보고지침의 저자, MR 연구설계를 자주 사용하는 연구자, 학술지 편집인 등으로 구성된 전문가 그룹을 워크숍에 초청하였다. 이 그룹은 2019년 5월 영국 브리스톨에서 이틀간 대면으로 만나 MR 연구의 보고 질에 관한 실증적 근거를 논의하고 체크리스트 초안을 마련하였다. 초안은 2019년 7월 프리프린트로 공개되었고[
29], 프리프린트 플랫폼, 소셜 미디어, 제4회 국제 멘델 무작위화 학술대회 전용 세션에서 논의되었다[
30]. 우리는 의견을 반영해 체크리스트를 수정하고 STROBE-MR 성명서를 제시하는 논문을 작성하였다[
27].
STROBE-MR 보고지침은 생식계열(germline) 유전변이의 특성을 이용해 조절 가능한 노출의 잠재적 효과가 결과에 미치는 인과추론을 강화하는 연구에 적용된다. MR의 주요 유형은 단일 표본(one-sample)과 두 표본(two-sample)이다. 단일 표본 MR에서는 유전변이–노출 관련성과 유전변이–결과 관련성을 동일 표본에서 모두 측정한다. 두 표본 MR에서는 두 관련성을 서로 다른 표본에서 측정한다. MR 연구는 단일염기다형성(SNP)별 가중치를 도출ㆍ적용할 때 개별 수준 자료 또는 요약 수준 자료를 사용할 수 있다. 두 표본 MR은 대개 요약 수준 자료로 수행되며, 첫 번째 표본에서 도출한 가중치를 두 번째 표본에 적용해 유전자–결과 관련성을 추정한다. 노출과의 관련성에 대한 요약 가중치는, 이들 변이와 결과의 관련성을 개별 수준 분석에 적용하는 데도 사용할 수 있다. STROBE-MR이 다루는/다루지 않는 연구설계 개요는 Box 1의
Table 1에 제시되어 있다.
이 논문의 목적
이 설명 및 해설(explanation and elaboration, E&E) 문서는 STROBE-MR 성명서를 보완한다[
27]. 형식은 STROBE E&E 문서[
26] 등 기존 보고지침을 따르며, 체크리스트 20개 항목 각각을 뒷받침하는 상세 설명과 투명한 보고 사례를 제공하는 데 목적이 있다. 각 항목의 우수한 보고 사례는 출판된 MR 연구에서 선정하였다.
이 문서는 저자가 체크리스트의 각 항목을 더 잘 이해하도록 돕는 참고자료로 보아야 한다. 제시한 예시는 각 항목의 ‘이상적’ 진술을 뜻하지 않으며, 해당 항목이 다루려는 쟁점을 부각하는 데 목적이 있다. 본문의 Box와 Table은 MR 연구설계에 관한 이론적 배경을 담아 보고 권고사항을 보완한다. MR 수행에 관한 추가 지침은 다른 자료에 제시되어 있다[
31].
일부 예시는 해당 항목과 무관한 인용•문단을 제거해 편집하였다. 항목은 제목ㆍ초록(항목 1), 서론(항목 2–3), 방법(항목 4–9), 결과(항목 10–13), 고찰(항목 14–17), 기타 정보(항목 18–20)로 구분된다(
Table 3). 일부 항목은 동일 주제의 하위 항목을 가진다(예: 항목 10d는 두 표본 MR에만 해당). 추가 예시는
Supplement 2에 있다. 지면 제약으로 일부 정보를 보충자료에 보고했더라도, 저자는 체크리스트의 모든 항목을 논문에서 다루는 것이 바람직하다.
제목과 초록(항목 1)
연구의 주요 목적이 MR일 때, 제목 및/또는 초록에 MR을 연구설계로 명시한다.
제목
예시
“BMI as a Modifiable Risk Factor for Type 2 Diabetes: Refining and Understanding Causal Estimates Using Mendelian Randomization [
32].”
“Genome Wide Analyses of >200,000 Individuals Identify 58 Loci for Chronic Inflammation and Highlight Pathways that Link Inflammation and Complex Disorders [
33].”
설명
MR이 연구설계에서 핵심적 역할을 하였다면, 제목에 “Mendelian randomization”을 포함하는 것이 바람직하다. 1차 분석이 MR이 아니고 MR이 후속 분석기법으로 사용된 경우에는 제목에 MR을 직접 포함하지 않고 논문의 주목적에 초점을 유지할 수 있다.
초록
예시
“Importance: 인간 유전 연구는 혈장 지단백(a)(Lp[a])가 관상동맥질환(coronary heart diseases, CHD) 위험과 인과적으로 관련됨을 시사해 왔다. 그러나 Lp(a)를 25%–35% 낮추는 여러 치료의 무작위배정시험은 Lp(a) 저하가 CHD 위험을 감소시킨다는 근거를 제공하지 못하였다.
Objective: LDL-C 1 mmol/L (38.67 mg/dL) 변화가 CHD 위험을 의미 있게 낮추는 효과와 동등한 근거를 보이기 위해 필요한 Lp(a) 농도 변화의 크기를 추정한다.
Design, setting, and participants: 5개 연구의 개별자료로 MR 분석을 수행하고, 48개 연구의 요약자료로 외부 검증을 하였다. 코호트ㆍ환자-대조군 연구에 포함된 개별자료는 CHD 20,793명과 대조군 27,540명이며, 요약자료는 CHD 62,240명과 대조군 127,299명을 포함한다. 분석기간은 2016년 11월–2018년 3월이다.
Exposures: 유전적 LPA 점수와 혈장 Lp(a) 질량 농도.
Main outcomes and measures: 관상동맥질환.
Results: 참가자의 53%는 남성이었고, 모두 백인 유럽계였으며, 평균 연령은 57.5세였다. 유전적으로 예측된 Lp(a)와 CHD 위험의 관련성은 Lp(a) 절대 변화에 선형적으로 비례하였다. 유전적으로 예측된 Lp(a) 10 mg/dL 감소는 CHD 위험 5.8% 감소(odds ratio [OR], 0.942; 95% confidence interval [CI], 0.933–0.951; P=3×10−37)와 관련성되었고, LDL-C 유전 점수로 추정한 LDL-C 10 mg/dL 감소는 CHD 위험 14.5% 감소(OR, 0.855; 95% CI, 0.818–0.893; P=2×10−12)와 관련되었다. 따라서 Lp(a) 농도 101.5 mg/dL 변화(95% CI, 71.0–137.0)가 LDL-C 38.67 mg/dL 변화와 동등한 CHD 위험 감소와 관련성되었다. 유전적으로 예측된 Lp(a)와 CHD 위험의 관련성은 스타틴, PCSK9 억제제, 에제티미브를 모사하는 변이에 의해 매개되는 LDL-C 변화와 독립적으로 보였다.
Conclusions and relevance: Lp(a) 저하의 임상적 이득은 Lp(a) 절대 감소량에 비례할 가능성이 크다. CHD 위험을 LDL-C 38.67 mg/dL 저하와 유사한 크기로 낮추려면 약 100 mg/dL 규모의 Lp(a) 절대 감소가 필요할 수 있다[
34].” (추가 예시는
Supplement 2 참조)
설명
초록은 수행 내용과 발견 내용을 균형 있게 요약해야 하며, 자료 출처, 노출ㆍ결과, 개별/요약자료 사용 등 설계의 핵심 이슈와 함께(가능하면) “Mendelian randomization”을 포함해 검색 용이성을 높이는 것이 바람직하다. 결과는 적용한 접근 전반에 걸쳐 점추정치와 불확실성(오차) 정보를 함께 제시해야 하며, P값만을 보고하는 것은 피한다. “인과적”이라는 표현은 신중히 사용해야 하며, MR이 특정 가정하에서 인과 관련성을 이해하도록 고안된 추정임을 분명히 해야 한다. 가능하면 구조화 초록을 사용해 관련 정보를 빠짐없이 담는 것이 좋다.
Supplement 2에는 단일ㆍ두 표본ㆍ내재형(embedded) MR 초록의 추가 예시가 있다.
서론
배경(항목 2)
보고하는 연구의 과학적 배경과 근거를 설명한다. 노출은 무엇인가? 노출–결과 간 인과성이 개연적인가? MR이 연구질문에 유용한 이유를 정당화한다.
예시
“역학 연구는 특히 여성에서 사춘기 연령이 이를수록 다발성경화증(multiple sclerosis, MS) 위험이 증가한다는 결과를 보고해 왔다. 그러나 이를 재현하지 못한 연구도 있다. 사춘기 시기는 체중 상태와 복잡하게 상호작용하며, 소아기의 높은 체지방은 이른 사춘기로 이어지고, 이는 다시 성인기의 높은 body mass index (BMI)와 관련된다. 증가한 BMI가 MS 병태생리에 기여한다는 근거가 있으므로, 관찰된 ‘사춘기 시기–MS’ 관련성의 일부는 BMI로 설명될 수 있다. 관찰연구의 일부 한계는 도구변수 방법으로 완화할 수 있는데, 이는 노출의 효과를 결과에서 탐색하기 위해 노출의 대리변수를 사용하는 접근이다. 멘델 무작위화(MR)에서는 유전변이를 도구변수로 사용해 위험요인과 결과 간 인과 관련성을 검정한다[
35].”
“비선형 관계는 의생명과학 전반에서 위험요인–결과 사이에 존재한다. BMI와 심혈관질환과 같은 노출 간 비선형 관계를 추정하는 방법이 개발되었다. 유전 성분을 제거한 BMI(도구변수 비포함 BMI)의 분위 오분위별로 BMI가 각 위험요인에 미치는 국소 평균 인과효과를 추정하고, 이 값들에 대해 이질성 및 추세 검정을 수행하였다[
36].”
설명
가설 없이 다양한 결과에 대한 노출의 효과를 탐색적으로 시험한 MR도 있으나[
37], 다수의 MR 연구는 선행 근거에서 도출된 구체적 가설을 평가하도록 설계된다. 특정 가설을 사용하는 경우, 현 가설을 평가하는 사전 기대 효과크기 포함한 근거를 제시해야 한다. MR은 인과 영가설 검정 또는 점ㆍ기간ㆍ평생 효과 추정에 쓰일 수 있다. MR이 연구가설을 평가하는 데 어떤 역할을 하는지 명확히 하여, MR 적용이 문헌의 어떤 공백을 메우는지 독자가 이해하도록 해야 한다.
사전 규정된 인과 가설(있다면) 포함하여 구체적 목적을 명확히 기술한다. MR이 특정 가정하에서 인과효과 추정을 목적으로 하는 방법임을 명시한다.
예시
“목적: 혈청 칼슘 상승과 관련된 유전변이가 관상동맥질환(coronary artery disease, CAD) 및 심근경색 위험과 잠재적 인과 관련성을 갖는지 멘델 무작위화를 이용해 평가한다[
38].”
설명
연구가 특정 노출이 특정 결과에 미치는 인과효과 추정을 목표로 함을 명확히 밝혀야 한다. 이 부분에서 관심 노출ㆍ결과를 정의해 독자가 맥락을 파악하게 하고, 전체 연구목적을 제시한다.
방법
연구설계와 데이터 출처(항목 4)
논문 서두에 연구설계의 핵심 요소를 제시하고, 연구의 모든 단계에 대한 데이터 출처를 나열한 표를 포함하는 것을 고려한다.
예시
“기여한 GWAS 컨소시엄의 세부는
Table 4에 제시하였다. 연구는 심혈관ㆍ대사 건강 관련 형질을 조사하고, 표본 수가 가장 크며, 표본 중복을 최소화하면서 인구 구성이 가장 유사한 것을 기준으로 선택하였다. 표본 중복 비율은
보충표 S1에 제시하였다. 주관적 웰빙은 행복ㆍ긍정정서ㆍ전반적 삶의 만족과 관련된 항목으로 측정하였다. 각 구성요소의 GWAS를 메타분석해 주관적 웰빙을 포착하였다. 모든 형질의 표현형 정의와 GWAS 방법에 관한 추가 정보는
보충표 S2를 참조하라. 혈압을 제외한 모든 표현형 점수는 z-점수로 표준화하였다[
39].”
설명
STROBE와 마찬가지로[
26], 논문 서두에서 연구설계의 핵심 요소를 제시하면 독자가 연구의 기본 구도를 빠르게 파악할 수 있다. 저자는 MR 연구가 개별 수준 참가자 자료를 사용했는지, 아니면 SNP 수준 요약자료를 사용했는지, 그리고 단일 표본설계인지 두 표본설계인지 명확히 해야 한다. 두 표본 MR에서는 한 단계는 요약자료, 다른 단계는 개별자료를 사용할 수도 있다. 일부 MR 연구는 여러 데이터 출처를 결합한다(예: 유전변이–노출 관련성은 한 출처, 유전변이–결과 관련성은 다른 출처). 또한 데이터 출처가 여러 표본의 메타분석일 수도 있다. 그러므로 전체 설계와 데이터 출처를 명확히 밝혀야 한다.
우리는 MR 연구에서 유전변이 수준 정보의 출처를 명확히 기록한 표(예:
Table 4)를 제시할 것을 권장한다. 예컨대 노출을 추정하는 데 사용한 유전변이는 한 연구에서 확인되었지만, 이 유전변이가 노출에 미치는 효과크기(또는 가중치)는 다른 연구에서 가져왔을 수 있다. 이런 경우 두 정보의 출처를 모두 보고하는 것이 바람직하다. 필요에 따라 표를 확장한다. 예를 들어, 서로 다른 결과를 갖는 추가 MR 연구를 포함한다면 표에 열을 더하고, 추가 노출을 다룬다면 행을 추가한다.
자료가 기존 연구에서 추출되었다면, 데이터가 어떻게 수집ㆍ획득되었는지 기술한다. 데이터가 공개 접근 가능하다면, 가능한 경우 출처의 하이퍼링크를 제공한다. 요약자료를 사용하는 경우, 이러한 세부가 추적 가능해야 하며 데이터 출처 간 이질성을 정성적으로 평가할 수 있어야 한다. 분석에 기여한 각 데이터 출처에 대해서는 항목 4a–4e의 요소를 기술한다.
환경(Setting, 항목 4a)
가능하면 연구설계와 기반 모집단을 설명한다. 가능하면, 장소, 위치, 관련 시기(모집, 노출, 추적, 자료수집 기간 포함)를 기술한다.
예시
“이 연구는 21개 코호트(총 42,024명)에서 직접 유전형 분석 및 보간(imputation)된 SNP를 대상으로 한 메타분석으로 구성되었다(
Table 1). 참여 연구의 확장 설명은
Supplement 2에 제시하였다.”[
40]
“전장유전체 유전형과 골절 자료를 보유한 총 23개 코호트가 GEFOS 컨소시엄(
http://www.gefos.org/)을 통해 전 세계에서 모집되었다. 이들 코호트는 주로 유럽계였으며, 유럽(n=13), 북미(n=8), 호주(n=1), 동아시아(n=1)에 분포했고(표 S1A, S2A), 골절 사례 20,439명과 대조 78,843명이 포함되었다[
41].”
설명
연구 인구, 환경, 위치에 대한 정보는 결과의 맥락과 일반화 가능성을 평가하는 데 필요하다. 환경 요인이나 치료 같은 노출은 시간이 지나며 변할 수 있고, 연구방법 또한 진화한다. 연구가 언제 수행되었는지, 참가자가 어느 기간에 모집ㆍ추적되었는지를 아는 것은 결과 해석에 필요한 역사 정보를 제공한다. 이러한 정보가 선행 논문에 이미 기술된 경우, 명확한 인용만으로 충분할 수 있다. 참가자의 조상(ancestry) 정보를 제공하면 잠재적 이질성과 일반화 가능성을 이해하는 데 도움이 된다. 기존 연구의 요약 수준 자료를 사용하는 경우, 자료 출처 간 환경의 이질성을 정성적으로 평가할 수 있도록 세부 사항을 추적 가능하게 제시해야 한다.
참가자(Participants, 항목 4b)
선정기준, 참가자 출처, 선정방법을 제시한다. 표본 수와, 본 분석 전에 검정력/표본 수 계산을 수행했는지 여부도 보고한다.
예시
“UK Biobank는 2006–2010년에 영국 전역에서 37–73세(99.5%는 40–69세) 성인 50만 명 이상을 모집하였다. 참가자는 설문 및 인터뷰로 인구학, 건강상태, 생활습관을 제공했고, 인체계측, 혈압, 혈액ㆍ뇨ㆍ타액 검체를 채취하였다. 자세한 내용은 다른 문헌에 기술되어 있다. 우리는 초기 UK Biobank 데이터셋에서 백인 영국계 120,286명을 사용했으며, 이 중 119,669명은 유전 자료와 BMI 및 키 측정값이 모두 유효하였다. 다른 인종 집단은 개별적으로는 검정력이 부족하여 포함하지 않았다[
42].” (추가 예시는
Supplement 2 참조)
설명
참가자와 표집방법에 대한 상세 기술은 결과의 적용 가능성을 이해하는 데 도움이 된다. 모든 선정기준, 참가자 출처ㆍ선정방법, 해당될 경우 추적방법, 그리고 모집방식을 제시한다. 선행 연구에 기술된 경우에는 명확한 인용으로 대체할 수 있다. 요약 수준 자료를 사용하는 경우에도 출처별 참가자 이질성을 정성적으로 평가할 수 있도록 세부 사안을 추적 가능하게 기술해야 한다.
환자-대조군 연구에서는 사례ㆍ대조 선택이 결과 해석의 핵심이며, 선택ㆍ방법이 타당성에 큰 영향을 준다. 일반적으로 대조군은 사례가 발생한 모집단을 반영해야 한다.
주요 분석 전에 표본수/검정력 계산을 수행하였다면 설계나 통계방법 부분에 보고한다. 연구 기획ㆍ해석에서 표본 수 정보는 필수다. 검정력은 효과 추정치의 원하는 정밀도에 적합한 검정력을 얻기 위해 필요한 표본크기에 대한 정보를 제공한다[
43,
44]. 검정력 분석을 수행할 경우 연구 수행 전에 실시해야 한다. 통계적 검정력은 실험 전 검정력 계산보다 가정이 적게 필요하므로, 추정치의 95% 신뢰구간을 검토함으로써 가장 잘 확인할 수 있다.
유전변이의 측정, 품질관리(quality control, QC), 선정을 기술한다.
예시
“유전형 분석은 Affymetrix UK Biobank Array로 수행하였다. 상염색체 분석은 HRC 보간 변이 중 MAF >0.05%, 소수대립유전자 수 >5, info 점수 >0.3, 하드 콜률(call rate) >0.95, Hardy-Weinberg P>1×10
−6의 고품질 변이(최대 13,977,204개)로 제한하였다.”[
45]
“다양한 비만 관련 위험요인에 대한 유전 표지는 유럽계 참가자에서 관심 위험요인과 관련된 SNP (P<5×10
−8)를 포함하였다. LD R²<0.1 기준으로 상관된 SNP는 제외하였다… 가닥이 모호한 SNP (A/T 또는 C/G)는 proxy snps R 패키지(유럽 인구)를 이용해 관련된 대체 SNP (R²>0.8)로 치환하거나, 소수대립유전자 빈도가 0.4를 초과하면 분석에서 제거하였다[
46].”
설명
유전형 확인 및 QC정보를 제공하면 사용된 유전변이의 질을 평가할 수 있다. 두 표본 MR에서는 종종 선행 논문의 보충자료를 인용해야 한다.
방법론 섹션에서는 분석에 포함된 특정 유전 변이체의 선정 및 포함기준을 명확히 설명해야 한다. 관심 노출(역 MR인 경우 결과)에 할당된 변이를 기술하고, 각 변이에 대해 rsID 또는 염색체 기반좌표를 제시하며, 선정 이유, 참조 패널을 명확히 한다. 선정 이유에는 관심 노출/결과와의 관련성 근거, 대리지표(proxy) 사용할 때, 관련성불균형(linkage disequilibrium) 특성 등이 포함될 수 있다. 강한 관련성불균형에 있는 변이를 다수 포함하면 인과효과 추정에 추가 정보를 주지 못할 수 있고, 상관구조를 고려하지 않으면 표준오차 추정에 편향을 유발할 수 있다[
47]. 독립 변이 선정 임계치(예: r²), 참조 패널, 조사 인구를 명시한다. 다만 특정 유전자 영역의 생물학적 관련성이 분명한 경우, 관련성불균형이 있어도 포함할 수 있으며, 이때는 해당 변이가 관여하는 생물학적 경로, 포함 r² 임계치, 상관구조 모형화 방법을 제시한다.
연구에서는 분석에 사용된 단일염기다형성(SNP)에 대한 품질 관리 매개변수 추정값도 제공해야 한다. 이 정보에는 정보 점수(대입 품질 지표), 콜률(특정 SNP에서 대립유전자가 확인된 개체 비율), 하디-와인버그 평형 검정 P값(대입 또는 유전자형 분석 문제, 집단 분화, 비무작위 교배를 나타낼 수 있음)이 포함된다.
두 표본 MR 분석에서 유전 변이체 관리 및 데이터셋 조화를 도출하기 위해서는 추가 정보가 필요하다. 이 정보에는 양 데이터셋에 동일한 변이체가 존재하지 않을 경우 대리 변이체를 식별하는 데 사용된 조건(예: 관련성 불균형 임계값), 가닥 정렬의 유무 및 처리 방법, 효과 대립유전자와 비효과 대립유전자의 방향 등이 포함된다. 관련성의 시간적 안정성, 표본 특이성 또는 생물학적 타당성과 같은 다른 측면들은 선택된 유전 변이체가 도구변수로 사용될 수 있는 타당성을 이해하는 데 도움이 될 수 있다.
질병의 평가와 진단기준(항목 4d)
각 노출, 결과, 기타 관련 변수의 평가방법과 질병 진단기준을 기술한다.
예시—연속형 노출/결과
“연구 결과변수는 WHR(1ㆍ2b단계), 엉덩이ㆍ허리둘레(2a단계), 신체 구획별 지방량(3단계)… WHR은 허리둘레/엉덩이둘레 비로 정의했고, 둘레는 Seca 200 cm 줄자를 사용해 센티미터로 추정하였다… 구획별 지방량은 전신 저선량 X선 스캔인 DEXA로 그램 단위로 측정했으며, Lunar Prodigy 팬빔 스캐너(GE Healthcare)를 사용하였다. 훈련된 담당자가 표준 영상ㆍ자세 프로토콜에 따라 스캔했고, 모든 이미지는 훈련된 한 연구자가 표준화 절차로 DEXA 경계를 교정하였다[
48].” (추가 예시는
Supplement 2 참조)
설명
분석에 사용한 핵심 노출ㆍ결과ㆍ혼란요인의 선택과 정의를 제시한다. 결과가 여러 개이거나 가설-비제한 접근(hypothesis-free)을 썼다면, 다중 검정을 처리하는 방법과 함께 명확히 밝혀야 한다. 메타분석의 경우 연구별 정의를 포함하거나, 선행 보고가 있다면 간단 요약+명확한 인용으로 대체할 수 있다. 독자는 사례 정의의 민감도ㆍ특이도를 고려해 적합성과 일반화 가능성을 평가할 수 있다.
윤리 승인과 동의(항목 4e)
해당될 경우, 윤리위원회 승인과 연구참여 동의의 세부를 제공한다.
예시
“모든 참가자로부터 사전 동의를 얻었고, 연구 프로토콜은 지역ㆍ기관 윤리위원회의 승인을 받았다[
49].”
설명
모든 연구자는 2013년 개정 헬싱키 선언을 준수해 사람 대상 연구를 기획ㆍ수행ㆍ보고해야 한다[
50]. 책임 윤리위원회의 승인과 동의 획득에 관한 정보를 제공해야 하며, 공개 데이터나 기존 연구의 데이터를 사용하는 경우에도 이를 명시한다. 연구가 원 윤리 승인 범위에 부합하고, 원 동의 조건을 위반하지 않도록 해야 한다.
본 분석의 세 가지 핵심 도구변수 가정(관련성, 독립성, 배제 제한)과 추가/민감도 분석에 필요한 추가 가정을 명시한다.
예시
“모든 MR 분석과 마찬가지로, 이 연구는 유전 도구가 관심 위험요인과 관련되고, 잠재 혼란요인과 독립이며, 대체 경로(다면발현)를 통해서가 아니라 위험요인을 통해서만 결과에 영향을 미친다는 가정을 전제하였다[
51].”
“추가로, MR-Egger 회귀의 기울기는 다면발현을 보정한 인과추정치를 제공할 수 있다… 이 접근의 중요한 조건은 SNP의 노출과의 관련성이 그 SNP의 결과에 대한 직접효과와 독립적이어야 한다는 것으로, 이전에 InSIDE 가정으로 불렸다[
52].”
설명
세 가지 핵심 가정을(이상적으로는 방법 파트에서) 명시하면 MR의 전제와 그 타당성 판단을 독자가 이해하는 데 도움이 된다. 가정은 연구 맥락에 맞는 직관적 언어로 풀어 쓰는 것이 바람직하다. 가정을 명확히 하면, 가정 점검과 민감도 분석의 설계 근거도 함께 제시된다.
도구변수 추정으로 효과추정치를 제시할 때는 네 번째 가정—보통 효과 균질성[
53] 또는 단조성[
54]—도 명시해야 한다. 많은 MR 연구는 전통적 도구변수 추정기(2SLS, Wald 등)를 보강하기 위해 다른 방법을 병행하며, 이 방법들의 고유 가정도 기술해야 한다. 예를 들어 MR-Egger 회귀[
55]나 가중 중앙값 추정[
56,
57]은 다수 변이를 포함할 때 보조적으로 쓰인다. Box 2,
Fig. 1, Box 3에서 도구변수 가정, 흔한 위반, 평가법을 더 자세히 제시한다.
Box 2. 멘델 무작위화(MR)의 범위와 STROBE-MR 체크리스트
핵심 IV 추정 가정과 추가 가정
대부분의 MR 연구는 노출이 결과에 미치는 인과효과를 검정하기 위해 세 가지 핵심 IV 가정—관련성, 독립성, 배제 제한—에 의존한다(
Fig. 1, Box 3) [
16]. 도구변수 접근으로 효과크기를 추정하려면 네 번째 가정, 보통 효과의 균질성(homogeneity)을 추가로 둔다[
16,
58-
60]. 균질성 가정은 단조성(monotonicity) 가정—위험 대립유전자 수가 증가해도 어떤 개인에서도 노출 가능성이 낮아지지 않는다는 가정—으로 대체할 수도 있으며, 이 경우 연구 집단의 한 부분집단에 대한 효과를 추정하게 된다[
61].
가정 위반
배제 제한은 때때로 “수평 다면발현 없음” 가정(Box 3)으로도 불리지만, 다음과 같은 여러 방식으로 위반될 수 있다: 유전자–노출 상호작용, 시간가변 노출, 도구와 관련된 노출 측정오차, 다성분 노출등[
59]. 독립성 가정에 대한 우려는 흔히 조상/인구 층화에 의한 교란에 초점이 맞춰지지만, 선택ㆍ콜라이더 편향, 가계(세대) 효과, 선택적 짝짓기(assortative mating)등으로도 위반될 수 있다[
62-
64]. 다수의 변이를 사용하는 분석에서는 이 가정들이 각 변이에 대해 성립해야 한다. 아래에 서술하듯, 일부 방법은 이러한 가정들을 완화할 수 있다.
추가 분석을 위한 가정
많은 MR 연구에서 도구변수 방법은 여러 방식으로 확장되어 왔다. 예를 들어 다수의 유전변이를 사용할 때, MR-Egger 회귀[
55], 가중 중앙값[
56], 가중 최빈값[
57] 추정기를 보조적으로 쓰는 경우가 흔하다. MR-Egger 회귀는 배제 제한가정을 완화하는 대신, 노출을 통하지 않는 직접효과의 크기가 변이의 노출 효과크기와 독립이어야 한다는 InSIDE 가정을 부과한다. 또한 두 표본 MR접근은 유전변이–노출 관련성이 두 표본에서 동일하다고 가정하는데, 표본이 성별ㆍ연령ㆍ민족 등 서로 다른 하위집단에서 추출된 경우에는 이 가정이 성립하지 않을 수 있다.
Box 3. 멘델 무작위화의 가정 평가와 민감도 분석
관련성(Relevance)
관련성 가정에 대해, 저자는 도구 강도를 어떻게 측정했는지 보고해야 한다. 개별 수준 자료가 있을 때 F 통계량을 보고하면 약한 도구 편향 위험을 이해하는 데 여러 이점이 있다[
65]. 요약수준 자료만 있어도 F 통계량을 근사할 수 있다. 제안된 도구 강도가 낮다면, 약한 도구에 강한 접근법을 사용했는지 여부를 함께 보고해야 한다.
독립성(Independence)
독립성 가정은 직접 검증할 수 없지만 많은 연구 맥락에서 부분적으로 점검할 수 있다. 음성 대조 결과나 음성 대조 집단을 사용해 가정의 타당성을 가늠할 수 있다[
66]. 변이–결과 관련성을 교란할 수 있는 측정된 공변량과의 관련성을 보고하는 것도 유익하며, 특히 도구 강도로 스케일링해 제시하거나 관련 편향 분석 기법과 함께 제시하면 도움이 된다[
67-
69].
배제 제한(Exclusion restriction)
배제 제한 가정의 경우, MR-Egger 회귀[
55]를 사용하면 특정 유형의 다면발현을 탐지할 수 있어, 배제 제한 위반의 정황증거를 제공한다. 다만 이 방법은 위에서 기술한 추가 가정(InSIDE)에 의존하고, 독립 변이 다수가 필요하다. 배제 제한을 점검하는 다른 접근으로 가중 중앙값[
56]과 가중 최빈값[
57] 방법이 있다. 음성 대조 결과/집단을 사용하는 것도 이 가정 평가에 도움될 수 있다[
70]. 아울러 SNP의 알려진 생물학적 효과를 활용해 이 가정의 위반 가능성을 낮출 수 있다.
균질성(Homogeneity)
균질성 가정은 모든 개인에서 노출의 효과가 동일하다는 것을 요구하며, 직접 검증할 수 없다. 이를 뒷받침할 하나의 가능성은, 효과 추정치(또는 유전변이의 노출에 대한 효과)가 하위집단 간에 동일한지 확인하는 것이다[
67,
71]. 서로 다른 하위집단에서 의미 있는 차이가 관측되면, 층화 분석이나 보정 분석으로 이 가정을 완화할 수 있다[
72]. 더 나아가, 연속형 결과의 분산이 유전 도구 수준에 따라 달라지는지 전반적으로 탐색해, 그 차이의 크기로 균질성 가정 위반의 정도를 가늠할 수 있다.
공동 반증 전략(Joint falsification strategies)
일부 반증 전략은 가정들을 공동으로 평가한다. 다수의 유전변이를 도구로 사용할 때는, 개별 변이별 효과추정치의 이질성이 존재하는지 시험할 수 있다(
Table 2의 ‘차이 검정’ 참조). 이 검정은 흔히 배제 제한 평가로 이해되지만, 실제로는 배제 제한, 독립성, 균질성을 동시에 시험한다. 또 다른 비교적 단순한 공동 검정은, 전통적 보정 접근으로 얻은 효과추정치와 MR 추정치를 비교하는 것이다[
73]. 전통적 접근이 미측정 교란으로 편향되었고 그 방향이 예상 가능하다고 가정하면, MR 추정치가 그 예상 방향과 어떻게 부합하는지를 점검함으로써 MR 추정치의 기초 가정들의 공동 타당성을 뒷받침할 수 있다.
민감도 분석(Sensitivity analyses)
다수 변이를 사용하는 여러 추정기는 도구변수 가정을 서로 다른 방식으로 완화/적응한다(예: MR-Egger, 중앙값 기반, 최빈값 기반추정기). 따라서 각 접근으로 얻은 효과추정치들을 비교하면, 각 방법의 서로 겹치지 않는 가정들에 대한 결과의 민감도를 파악할 수 있다[
74]. 필요에 따라 MR 추정치와 비-MR 추정치를 비교할 수도 있다. 결과의 견고성을 평가하기 위해, 독립 데이터셋이나 다른 설계(단일 표본, 두 표본)로 독립 재현을 수행하는 것을 권장한다.
역학의 전통적 편향 분석 기법도 MR에 쉽게 응용할 수 있다. 예컨대 교란 편향의 크기와 방향을 이해하기 위한 공식 기반 계산[
59,
67-
69,
75], 배제 제한 위반을 정량화하는 절차[
59,
76] 등이 있다. 선택 편향이 우려될 때는 가능한 편향의 크기•방향을 이해하기 위해 시뮬레이션을 자주 수행한다[
64]. 또한 선택적 짝짓기[
62], 가계(세대) 효과[
9,
63], 시간가변 효과[
77]로 인해 유발될 수 있는 편향의 크기ㆍ방향을 파악하는 데도 시뮬레이션이 유용하다.
통계방법: 본 분석(항목 6)
사용한 통계방법과 통계치를 기술한다.
정량 변수(항목 6a)
분석에서 정량 변수를 어떻게 다루었는지(척도, 단위, 모형)를 기술한다.
예시
“각 메타분석의 효과크기는 주결과에서 25OHD[25-하이드록시비타민 D] 농도의 자연로그 변환 값 1표준편차(1-SD) 변화에 대한 효과로 보고하였다. 이 지표가 임의 차이보다 해석에 더 적합하기 때문이다… 1-SD 로그변환 25OHD 변화의 임상적 해석을 돕기 위해 비타민 D 상태의 세 임상 역치(<25 nmol/L 결핍, <50 nmol/L 불충분, >75 nmol/L 충분)를 선택하였다[
78].”
설명
정량 변수(노출, 결과, 관련 공변량)에 가한 변환은 결과 해석과 타 연구와의 비교가능성에 영향을 주므로 명시해야 한다. 생물학적 지식이나 선행 근거를 근거로 군집화ㆍ범주화의 타당성을 제시하라. 가능하다면 추정치를 역변환해 일반적 단위로도 보고해 재현을 돕는다. 예를 들어, 효과 크기를 표준편차 변화로 보고하는 경우 명확성을 위해 표준편차의 크기를 함께 보고할 것을 권장한다.
유전변이(항목 6b)
유전변이를 분석에서 어떻게 다루었는지, 해당 시 가중치를 어떻게 선택했는지 기술한다.
예시
“우리는 GIANT 컨소시엄의 최신 GWAS 메타분석에서 BMI와 관련된 97개 유전변이를 기반으로 대립유전자 점수를 만들었다. 점수는 BMI-증가 대립유전자 개수의 합을, GIANT GWAS에서 보고된 효과크기(대립유전자 용량 1단위 증가당 BMI의 SD 변화)로 가중해 계산했고, 평균 0, SD 1이 되도록 표준화하였다. 점수가 높을수록 BMI가 높다[
79].”
설명
대립유전자 점수(=유전위험점수, 다유전자 점수, 유전 예측점수)는 여러 변이를 하나로 요약한 변수다. 점수에 많은 변이를 포함할 경우, 도구변수 추정값의 편향 및 포괄 확률(coverage probability)은 2단계 최소제곱법 접근법(2 stage least squares approach)으로 얻은 추정값에 비해 개선된다[
80]. 점수에 포함할 변이의 선정기준(외부 데이터 기반 여부 포함)을 명시한다. 점수는 가중/비가중일 수 있으며, 가중치가 있다면 동일 표본 산출인지 또는 독립 표본에서 가져왔는지 밝힌다. 변이–노출/변이–결과 관련성을 추정할 때 어떤 유전 모형(가산/승산)을 가정했는지도 보고한다. 가중치를 같은 표본(예: 단일 표본 MR)에서 추정하였다면, 과적합을 줄이기 위한 교차검증ㆍ잭나이프(jackknife) 등 절차를 보고한다.
MR 추정량(항목 6c)
사용한 MR 추정량(예: 2단계 최소제곱, Wald 비)과 관련 통계를 기술한다. 포함 공변량을 상세히 적고, 두 표본 MR이라면 두 표본에서 동일 공변량을 사용했는지도 보고한다.
예시
“모든 노출에 대한 유전 관련성은 당뇨병이 없는 유럽계 성인(n=108,557; 평균 50.6세; 남성 약 53%)을 대상으로 한 대규모 GWAS 메타분석에서 가져왔고, 연령ㆍ성별ㆍ연구지ㆍ지리 공변량을 보정한 가산 유전 모형을 사용하였다… MI(심근경색), 협심증, 심부전에 대한 유전 관련성은 성별별 분석에서는 연령ㆍ칩 종류ㆍ주성분 10개를 보정한 로지스틱 회귀, 전체 분석에서는 여기에 성별을 추가 보정하여 추정하였다. SNP별 Wald 추정치(결과에 대한 유전 관련성÷인슐린에 대한 유전 관련성)를 구한 뒤, 가법 랜덤효과 IVW로 메타분석하였다[
81].”
설명
도구변수 추정량의 계산과 표준오차추정(정규근사, 부트스트랩 등)을 명확히 제시해야 한다. MR 분석에 사용한 공변량을 상세히 쓰고, 두 표본 MR에서는 변이–노출과 변이–결과 관련성의 보정 공변량 차이가 편향을 유발할 수 있음을 고려하여 이를 보고해야 한다.
결측자료(항목 6d)
결측자료를 어떻게 다뤘는지 설명한다.
예시
“분석은 베이지안 체계에서 수행해 자료 대치가 자연스럽게 가능하도록 하였다. 먼저 베이지안 완전사례 분석방법을 제시하고, 무작위결측 가정하에서 베이지안 모형에 통합 가능한 4가지 대치방법을 제안하였다… 우리는 CRP, 피브리노겐, 3개 SNP의 완전 또는 부분 자료가 있는 3,693명의 단면 기저선 자료를 사용하였다. 결측은 CRP 2.1%, 피브리노겐 2.4%, rs1205 10.8%, rs1130864 1.9%, rs1800947 2.6%였다[
82].”
설명
여러 변이에 결측이 있으면 인과효과 추정의 정밀도가 저하될 수 있다. 변수별 결측 비율과 대치 수행 여부, 대치 시 패널과 방법을 보고한다.
다중 검정(항목 6e)
해당하는 경우, 다중 검정을 어떻게 처리했는지 명시한다.
예시
“모든 암의 위험•사망에 대한 유의수준은 0.004로 설정하였다(6개 PUFA×2개 결과이므로 0.05를 12검정으로 보정)… 6개 개별 암을 추가로 고려할 때는 유의수준을 0.004/36=0.0001로 설정하였다[
83].”
설명
다중 노출 또는 다중 결과를 포함하는 MR 분석에서 저자는 다중 검정 문제를 고려했는지 여부와 그 방법을 명시하고 그 근거를 제시해야 한다. 또한 통계적으로 독립적인 모든 노출 변수 또는 결과 변수에 대한 총 검정 횟수에 대한 보정이었는지 여부를 명시해야 한다. 이러한 보정에는 위 예시에서 설명한 바와 같이 거짓 발견률(false discovery rates), 본페로니 보정 또는 기타 기법의 보고가 포함될 수 있다.
가정의 평가(항목 7)
가정을 평가하거나 타당성을 정당화하기 위해 사용한 방법 또는 선행 지식을 기술한다.
예시 1
“MR은 R 패키지 ivpack의 2단계 최소제곱법으로 구현하였다. 공변량으로 연령과 성별을 포함하였다. 약한 도구 편향 위험을 평가하기 위해 1단계 회귀에서 대립유전자 점수–노출 관련성의 F 검정을 사용하였다… 표준 다변량 회귀와 비교해 도구변수 추정의 상대 편향을 평가하기 위해 confounding bias plot을 사용하였다… 초기 인과추정의 다면발현에 의한 편향 정도를 조사하기 위해 MR-Egger와 가중 중앙값 MR 두 가지 민감도 분석을 수행하였다… MR-Egger와 가중 중앙값 방법은 R 패키지 TwoSampleMR로 구현하였다[
84].”
예시 2
“단백질 약물표적에 대한 MR은 구별되는 범주로 볼 이유가 있다… mRNA 발현을 제외하면, 단백질 발현이나 기능의 차이가 자연 유전변이의 가장 근접한 결과다. 이로 인해 표적 유전자 주변 변이는 다른 형질과 비교해 단백질 발현에 큰 효과를 보일 수 있고, 또한 전장유전체 다른 위치의 변이보다 수평 다면발현가정 위반에 덜 취약할 수 있다… 단백질 MR의 경우 크릭의 ‘중심원리’(gene→mRNA→protein)가 유전정보의 흐름 방향을 규정하며, 이는 질병 위험으로 이어지는 인과사슬의 더 먼 생물학적 형질에는 확장되지 않는다. 마지막으로, cis-MR은 유전자→암호화 단백질→질병 경로가 유전자→질병→단백질 경로보다 항상 우세하다는 점에서 역인과위험을 크게 줄인다(대개 유전자→단백질 관련성은 질병 없는 모집단에서 얻어지기 때문). 따라서 MR 관점에서 단백질은 다른 범주의 위험요인보다 특권적 위치에 있고, cis-MR은 질병에 대한 단백질의 인과효과를 도구화하는 최적 접근을 제공한다[
85].”
설명
MR의 각 기초 가정에 대해, 이를 평가하거나 타당성을 뒷받침하기 위해 사용한 방법을 보고해야 한다. 주제 관련 배경지식으로 가정의 개연성을 뒷받침할 수 있다. 많은 가정은 검증은 어렵지만 반증을 시도할 방법이 있다. 관련성 가정에 따라, 저자들은 도구 강도를 어떻게 평가했는지 보고할 수 있다. 가능한 방법은 많지만 일부는 특정 상황(예: 이분형 노출)에만 적용 가능하다. Box 3은 흔하고 유용한 접근을 요약하고,
Table 5는 가정 점검과 민감도 분석에 흔히 쓰이는 통계를 정리하였다. 처음 세 가지 핵심 가정은 단일 도구MR에도 해당하며, 도구변수 추정을 위해서는 추가 가정이 필요하다. 배제 제한은 MR-Egger와 같은 민감도 분석에서 완화될 수 있다.
이들 평가는 가능한 전략의 전부가 아니며, 모든 MR에 모든 민감도 분석이 필요한 것도 아니다. 예를 들어 F 통계는 본질적으로 도구변수 분석에서 중요하며, GWAS 산출물 기반 두 표본 MR에서는 설명분산과 근사적으로 일치한다. 측정된 공변량(연령, 성별, 인종/민족)과 하위집단별 효과추정은 단일 표본 MR에서 보고할 수 있으나, 두 표본 MR에서는 일반적으로 어렵다. 다만 성별ㆍ조상 특이 GWAS 요약통계가 점점 더 이용 가능해지고 있다. 더 포괄적인 검토는 Glymour 등[
73], Labrecque와 Swanson [
86]을 참조한다.
수행한 민감도 분석 또는 추가 분석(예: 서로 다른 접근 간 효과추정 비교, 독립 재현, 편향 분석 기법, 도구 검증, 시뮬레이션 등)을 기술한다.
예시
교란(Confounding): “우리는 표준 다변량 회귀와 비교해 도구변수 추정의 상대 편향을 평가하기 위해 confounding bias plot을 사용하였다. 이 분석은 표준 회귀에서 특정 교란변수를 보정하는 경우와 보정하지 않는 경우의 차이를 살피듯, MR 분석에 내재한 편향의 크기를 정량화하도록 고안되었다. 추가로 보충분석에서 의심 교란요인을 공변량으로 포함하였다(
Table 4). 고려한 교란변수는 유전 주성분 1–10, Townsend 박탈지수, 출생체중, 모유수유 여부, 출생지(북위ㆍ동경 좌표)였다.”
수평(유전) 다면발현: “초기 인과추정의 다면발현에 따른 편향 정도를 확인하기 위해 MR-Egger와 가중 중앙값 MR 두 가지 민감도 분석을 수행하였다. MR-Egger는 도구–노출 관련성과 도구–결과 관련성을 같은 표본에서 계산한 연구(본 연구 주분석과 같음)에는 타당하지 않다. 따라서 표본을 임의로 반분해 ‘A/B’ 분할 표본 분석으로 MR-Egger를 수행하였다. 보충표에는 각 군에서 변이와 교육기간, 굴절이상의 관련성이 제시되어 있다. MR-Egger와 가중 중앙값 방법은 R 패키지 TwoSampleMR (
https://github.com/MRCIEU/TwoSampleMR)로 구현하였다.”
측정오차: “교육을 마친 연령 변수의 비정규 분포가 ‘교육기간–근시’ 관련성을 인위적으로 만든 것이 아님을 확인하려고, 교육기간을 두 가지 방식으로 재부호화하였다: (1) 교육 종료 연령 >16세 vs. ≤16세로 이분화, (2) 대학(교)진학자 제외. 그런 다음 원래의 연속형 연령 변수를 쓴 분석과 결과를 비교하였다[
87].”
다른 추가 분석 예시: “개별 변이 관련성 검정은 유전자 단위 검정과 S-PrediXcan 분석으로 보완하였다. 후자는 대마 사용군과 비사용군의 차등 유전자 발현을 확인하는 데 사용하였다. 또한 평생 대마 사용과 다른 형질(다른 물질 사용, 조현병 등 정신건강 형질) 간 유전 상관을 추정하였다. 마지막으로, 쌍방향 두 표본 MR을 수행해 ‘대마 사용→조현병 위험’과 ‘조현병 소인→대마 사용’의 인과 방향성에 대한 근거를 검토하였다[
88].” (추가 예시는
Supplement 2 참조)
설명
민감도 분석은 기저 가정의 현실적 위반에 대해 견고성을 시험하고, 가능한 편향의 크기ㆍ방향을 가늠하게 해준다. 수행한 민감도 분석은 모두 보고해야 한다. 흔한 전략은 Box 3에 정리되어 있고, 추가 정보는 다른 자료에 제시되어 있다[
55,
74,
86].
통계 소프트웨어(항목 9a)
사용한 통계 소프트웨어와 패키지(버전ㆍ설정 포함)를 명시한다.
예시
“분석은 Stata v14 (StataCorp LP)와 R v3.4.3 (R Foundation)으로 수행하였다. MR 분석에는 Stata의 mrrobust패키지와 R의 TwoSampleMR 패키지를 사용하였다[
89].”
설명
통계방법ㆍ소프트웨어는, 원자료에 접근 가능한 숙련 독자가 결과를 재현 검증할 수 있을 정도의 세부로 기술하는 것이 바람직하다. 가능한 경우, 사용한 분석코드를 온라인 저장소에 제공하는 것이 좋다.
사전등록(항목 9b)
연구 프로토콜의 사전등록 여부(언제ㆍ어디에)를 명시한다.
설명
사전등록을 하였다면 그 사실과 프로토콜 링크를 제시해야 한다. MR에서 사전등록은 아직 드물며, 2차 자료 분석의 특성상 어려움이 있다. 연구자 편향을 줄이기 위한 대안으로, “연구의 근거ㆍ가설ㆍ방법ㆍ분석계획을 사전 명세하고, 이를 제3자 레지스트리(예: OSF,
https://osf.io/)나 학술지 Registered Report 형식으로 제출”하는 방안이 제안되어 왔다[
90]. 이러한 방식의 넓은 채택은 MR 연구의 정확성ㆍ투명성ㆍ견고성을 높일 것이다.
결과
서술적 자료(항목 10)
참가자 수(항목 10a)
포함 연구의 각 단계에서 개체 수와 제외 사유를 보고한다. 흐름도 사용을 고려한다.
예시
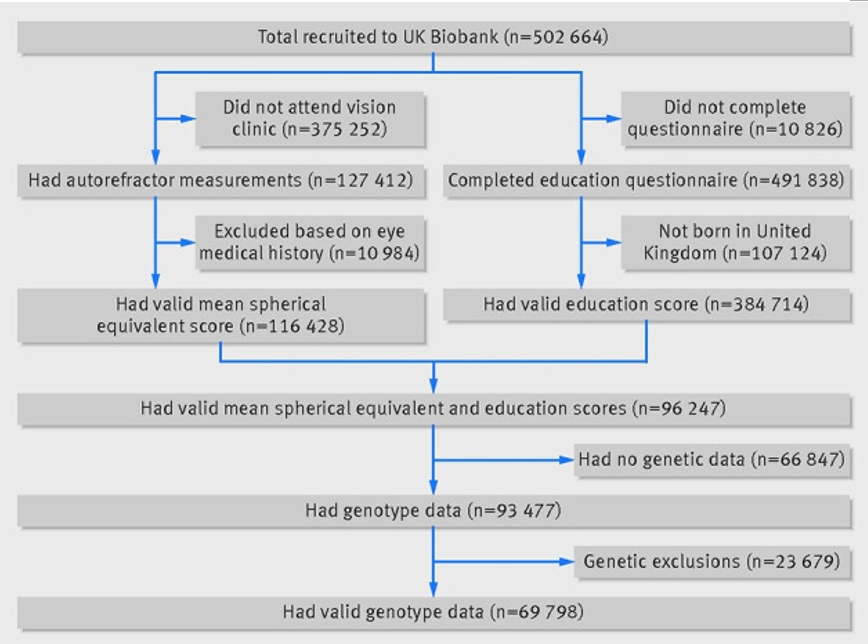
“UK Biobank는 영국 전역 22개 평가센터에서 40–69세 502,664명을 모집하였다… 모든 참가자는 과거 교육ㆍ직업 자격을 포함하는 사회인구학 설문을 완료하였다. 모집 후반에는 안과 평가가 도입되었고, 전체의 약 23%가 이를 완료하였다… 최종적으로 69,798명이 유효한 교육, 굴절이상, 유전자료를 보유하였다[
87] (
Fig. 2).”
설명
연구 참가자 정보는 표적 모집단을 이해하고 결과의 타당성과 일반화 가능성을 평가하는 데 도움이 된다. 또한 연구 재현에 필요한 정보를 제공하고, 연구가 콜라이더 편향을 보일 가능성이 있는지 판단하는 데 유용하다. 데이터 출처에 개별 수준 자료가 포함되어 있다면, 저자는 연구에 포함된 참가자 정보를 보고해야 한다. 구체적으로, 연구 각 단계의 개인 수와 추가 분석에서 제외된 사유를 제시한다. 제외 사유의 예로는 추적 손실, 자료 부재로 인한 제외, 품질관리(QC) 등이 있다. 연구 표본이 어떻게 선정되었는지 신속히 보여 주기 위해, 가능한 경우 STROBE 흐름도를 포함하는 것이 좋다[
26]. 또한 가능하면 변수별 결측값을 보고한다.
요약 통계(항목 10b)
표현형 노출, 결과, 기타 관련 변수에 대한 요약 통계(예: 평균, 표준편차, 비율)를 보고한다.
예시
“UK Biobank 표본은 여성이 53.7%였고(
Table 1), 모집 당시 중앙연령은 58.0세(사분위범위, 51.0–63.0)였다. UK Biobank 표본에서 체지방(노출)과 흡연행태(결과) 변수의 분포는
Table 6과
Table 7에 제시하였다. 선행 연구와 같이, 현재 흡연자는 비흡연자에 비해 BMI가 낮았고(−0.22; 95% 신뢰구간, −0.27 to −0.16), 반대로 과거 흡연자는 현재 흡연자보다 BMI가 높았다(1.04; 95% 신뢰구간, 0.98 to 1.09) [
51].”
노출, 결과, 그리고 기타 변수의 분포에 대한 정보는 집단 간 비교 가능성과 연구결과의 일반화 가능성을 판단하는 데 도움이 된다. 연속형 변수의 분포는 평균과 표준편차로 쉽게 요약할 수 있으며, 분포가 비대칭인 경우에는 중앙값과 분위수 범위(예: 25백분위수와 75백분위수)로 제시하는 것이 적절하다. 범주형 변수는 수와 백분율로 기술하는 것이 가장 적합하다. 기술통계는 각 범주별로 따로 제시할 때 독자가 집단 차이를 더 잘 평가할 수 있다. 집단 간 차이에 대한 통계적 추론은 본 분석에서 다루는 것이 바람직하다[
26].
코호트 연구에서 결과가 사건(event)일 때, 연구자는 사건 수와 필요하다면 사건률(예: 인년당 사건 수)도 함께 보고해야 한다. 또한 평균, 중앙값, 총 추적기간 등 추적기간의 요약지표를 제시하여 사건이 기록된 기간을 이해할 수 있게 하는 것이 중요하다.
시간에 따라 변하는 결과(time-varying outcome)로 생존시간 자료가 있을 때는, 요약지표를 시간에 따라 제시해야 하며, 그림을 활용하면 전달에 도움이 된다. 환자-대조군 연구에서는 보통 요약지표를 환자군과 대조군에 분리하여 제시한다. 연속형 노출이나 결과를 범주별로 나누어 표로 제시하는 것도 유용할 수 있다[
26].
이질성 평가(item 10c)
자료원에 기존 연구의 메타분석이 포함되어 있다면, 연구들 간 이질성에 대한 평가결과를 제공해야 한다.
예시
Table 8은 I² 검정 통계량을 제시하여, 유전 변이가 결과에 미치는 효과의 이질성을 평가할 수 있게 한다.
설명
유전변이와 노출 또는 결과의 관련성이 일관되는지에 대한 근거는 효과의 이질성 정도를 이해하는 데 도움이 된다. 추정이 메타분석에 기반한다면, 포함된 연구 수를 함께 제시하여 이질성 검정이 그 존재를 탐지할 충분한 검정력을 가졌는지 판단할 수 있게 하는 것이 바람직하다. I² 통계와 함께 95% 신뢰구간을 제시하는 것을 권장한다[
91,
92].
두 표본 멘델 무작위화(two-sample MR) (항목 10d)
두 표본 MR에서는 (1) 노출 표본과 결과 표본에서의 “유전변이–노출” 관련성이 유사하다는 근거를 제시하고, (2) 두 표본에 동시에 포함된 개인 수(노출 기준, 결과 기준)를 보고해야 한다.
예시 1
“MR에 사용된 유전변이는 유럽인에서 수행된 담석증 GWAS에서 얻었다. 인도 인구와 유럽 인구 간에 해당 변이들의 대립유전자 빈도, 담석증 및 담낭암(gallbladder cancer, GBC) 위험을 비교했으며, 결과는
보충표 1에 제시하였다. 두 인구의 대립유전자 빈도는 전반적으로 유사하였으나, 일부 SNP에서는 뚜렷한 차이를 보였다(예: rs601338, rs1260326, rs174567, rs2469991, rs2290846; 소수 대립유전자 빈도 차이 >15%). 인도 인구에서의 담석증 및 GBC 위험 방향은 대체로 유사했으며, 담석증 관련 SNP의 80%, GBC 관련 SNP의 70%에서 위험 증가 방향이 일치하였다[
93].”
설명 1
두 표본 MR 분석은 두 표본에서의 SNP–노출 관련성이 유사하다는 가정을 전제한다. 이는 두 표본이 동일한 기반 모집단에서 추출되었다는 가정과 유사하다. 다만 인종과 같은 특성만이 유일한 고려 요소는 아니다. 예를 들어 폐경 전 여성과 폐경 후 여성 간, 또는 지역사회 기반 표본과 고위험 표본 간에 유전 관련성이 추정되었다면, 표본 유사성이 깨질 수 있다. 이러한 가정을 단정하기 어렵다면, 가능한 경우 두 표본에서의 SNP–노출 및 SNP–결과 관련성을 비교하여 평가해야 한다. 두 표본에서 관련성이 유사하다면, SNP–노출/결과 관련성의 이질성이 편향을 일으킬 가능성은 낮아진다. 또한 저자는 노출과 결과 각각에 대해 두 표본에 중복 포함된 개인 수를 보고해야 한다.
예시 2
“이들 GWAS 추정치는 참가자 중복을 피하기 위해 UK Biobank 참여자를 포함하지 않은 연구에서 선택하였고, 그 결과 앞서 기술한 두 표본 멘델 무작위화에 사용된 GWAS 및 도구 변수가 일부 경우에서 상이하였다[
89].”
설명 2
동일하거나 유사한 개인을 사용해 SNP–노출과 SNP–결과 관련성을 동시에 추정하면, 발견 표본에서 통계적으로 가장 강한 관련성(대개 P값 기준)을 선택하는 과정에서 ‘승자의 저주’로 인한 편향이 발생할 수 있다[
94,
95]. 이 편향은 SNP 선택과 SNP–결과 추정을 완전히 분리된 표본에서 수행함으로써 줄일 수 있다. 편향의 크기는 두 집단에 중복 포함된 개인 수에 선형적으로 비례하므로, 중복이 소량이면 영향이 크지 않을 수 있다[
94].
유전변이 관련성(항목 11a)
유전변이와 노출, 그리고 유전변이와 결과 간의 관련성을 해석 가능한 척도로 보고하는 것이 바람직하다.
예시
“12개의 BMI 관련 SNP로 구성한 BMI 대립유전자 점수는 BMI와 용량-반응 양의 관련성을 보였다(점수 1단위 증가당 0.14% [0.12%–0.16%], P=6.30×10
−62). 이 점수는 25(OH)D 농도와도 관련되었다(점수 1단위 증가당 −0.06% [−0.20% to −0.02%], P=0.004) [
40].”
설명
유전변이와 노출 간 관련성 보고는 관련성 가정(항목 8b)을 평가하는 데 필수이다. 유전자형 분포에 따른 노출 수준 비교는 유전 효과의 단조성과 선형성을 가늠하는 데도 도움이 된다. 유전변이와 결과의 관련성 보고는 노출-결과 간 인과 관련 가능성에 대한 초기 단서를 제공할 수 있다.
멘델 무작위화 추정치(항목 11b)
노출과 결과 간 관련성에 대한 MR 추정치와 불확실성 지표를, 표준편차 1단위당 오즈비 또는 상대위험도처럼 해석 가능한 척도로 보고한다.
예시
“유전적으로 예측된 BMI가 1표준편차 증가할 때 관상동맥질환(coronary artery disease, CAD)의 오즈비는 1.49 (95% CI, 1.39–1.60)였다[
96].”
설명
도구변수 가정이 반증되지 않고 전반적으로 지지되거나(항목 8b), 민감도 분석이 가정 위반에 견고하다면, MR 추정치는 상대위험도나 위험차 등 직관적 척도로 의미 있게 보고할 수 있다. 반면 균질성 및 단조성 가정이 성립하지 않으면, 추정치 보고를 지양하고 비영(非零) 효과에 대한 검정으로 대체하는 것이 바람직할 수 있다.
절대위험 계산(항목 11c)
적절하다면, 상대위험 추정치를 임상적으로 의미 있는 기간의 절대위험으로 변환하여 제시하는 것을 고려한다.
예시
“NPC1L1 좌위의 LDL 콜레스테롤 저하 대립유전자는 관상동맥질환과 역의 관련성을 보였다(유전적으로 예측된 LDL-C 1 mmol/L [38.7 mg/dL] 감소당 OR, 0.61; 95% CI, 0.42–0.88; P=0.008). 반면, 제2형 당뇨병과는 개별-집합 모두에서 양의 관련성을 보였다(동일 감소당 OR, 2.42; 95% CI, 1.70–3.43; P<.001; 절대위험차 추정치, 1,000인-연당 5.3건 증가) [
97].”
설명
기저위험을 고려하면, 상대위험보다 절대위험(또는 위험차)으로 제시하는 편이 임상적으로 더 해석 가능할 때가 있다. 절대위험은 특정 기간 동안 노출로 귀속 가능한 초과질환량을 추정하게 하며, 이를 통해 노출을 줄이는 중재의 절대 이득을 산출할 수 있다.
결과 시각화(항목 11d)
포레스트 플롯, 유전변이–노출 관련성 대비 유전변이–결과 관련성 산점도 등 도표를 활용해 결과를 시각화하는 것을 고려한다[
98] (
Fig. 3).
예시
설명
도구변수 가정 위반 가능성을 살피는 데 도표가 유용하다. 특히 배제 제한(exclusion restriction) 가정 점검에 도움이 된다. 저자는 유전변이별로 노출 및 결과와의 관련성을 각각 보고해야 하며, 산점도나 퍼널 플롯(funnel plot)으로 제시할 수 있다[
55]. 산점도는 유전효과와 노출의 관련성 대비 유전효과와 결과의 관련성을 나타내며, 직선의 기울기는 추정된 인과효과를, 절편은 원점에 고정된다(단, MR-Egger 회귀는 예외; 항목 8b). 퍼널 플롯은 변이별 인과효과 추정치를 정밀도에 대비해 그려 비대칭성을 시각적으로 점검하는 데 쓰이며, 비대칭성은 수평 다면발현의 단서일 수 있다[
55]. 각 유전변인으로부터 얻은 인과추정치를 나열하는 포레스트 플롯은 전체 인과추정치 주변의 이질성을 시각적으로 점검하게 한다[
84].
가정의 타당성(항목 12a)
가정의 타당성 평가결과를 보고한다.
예시—관련성(relevance) 가정
“근시 대립유전자 점수는 영국 바이오뱅크 참가자의 평균 구면 굴절오차 분산의 4.32% (F=3155)를 설명했고, 교육 대립유전자 점수는 교육에 사용한 시간의 분산 0.71% (F=464)를 설명하였다. 두 점수는 각각 교육시간과 근시와의 강한 관련성을 보여, 멘델 무작위화 추론에 쓸 수 있는 강한 집합 도구변수로 구성할 수 있었다. 큰 F 통계량은 약한 도구 편향이 문제되지 않을 것임을 시사하였다[
87].”
예시—독립성(independence) 가정
“교육시간과 근시에 대한 대립유전자 점수와 잠재 혼란요인 간 관련성을 시험한 결과, 영국 북향 거리(northing) 좌표는 교육시간과 음의 관련성(β=−1.6e−6; 95% CI, −1.8e−6 to −1.5e−6), 굴절오차와 양의 관련성(β=1.2e−6; 95% CI, 9.8e−7 to 1.3e−6)을 보였다. 노딩은 교육시간(P=7e−5)과 근시(P=6e−3) 대립유전자 점수와도 관련되었다(
보충표 2 참조). 표준 회귀와 비교했을 때, 혼란 편향 플롯은 도구변수 분석에 노딩을 포함하면 교육 점수에서는 더 큰 편향을, 근시 점수에서는 그렇지 않을 것을 시사하였다[
87].”
예시—배제 제한(exclusion restriction) 가정
“MR-Egger, 가중 모드, 가중 중앙값 방법은 모두 크기와 방향에서 유사한 인과추정치를 보였고, 교육시간이 증가할수록 근시 굴절오차가 더 근시 방향으로 이동하였다(−0.17 to −0.40 디옵터/년). 반면, 근시가 교육시간을 늘린다는 근거는 적었다… Egger 절편은 교육→근시(절편=0.007, SE=0.006, P=0.2)나 근시→교육(절편=−0.002, SE=0.007, P=0.8) 모두에서 0과 유의하게 다르지 않았고, 방향성 유전 다면발현의 근거가 적음을 시사하였다[
87].”
예시—균질성(homogeneity)
“유전적으로 예측된 BMI와 전체 사망률 사이에서 J자 형태의 관계를 관찰하였다. 이 곡선은 영국 바이오뱅크에서 더 뚜렷했고, 저체중과 과체중/비만에서 위험이 높았다. 전체 인구에서 최소 위험은 HUNT 연구에서는 BMI 약 22–23 kg/m
2, 영국 바이오뱅크에서는 약 25 kg/m
2였다[
99].”
설명
저자는 항목 7과 Box 3에서 기술한 바와 같이 도구변수 가정의 타당성 평가결과를 보고해야 한다. 위 예시는 가능한 평가의 일부일 뿐이며, 모든 평가 또는 가정을 포괄하지는 않는다.
추가 통계(항목 12b)
이질성(I², Q 통계량)이나 E-value 등 추가 통계를 보고한다.
예시
“이질성(각 SNP 효과의 분산) 존재 여부를 점검하기 위해 Cochran의 Q와 I² 통계를 계산하였다. BMI와 웰빙 간 관련성에서는 이질성의 근거가 적었다(자세한 내용은 보충표 S8) [
39].”
“Chen 등은 ALDH2 유전자의 단일 변이를 이용해 음주가 고혈압 위험에 미치는 영향을 연구하였다. 남성에서 변이–고혈압 관련성에 대한 오즈비는 2.42였고, E-value는 4.27이었다. 신뢰구간 하한(1.66)에 대한 E-value는 2.71이었다. 분석이 민족적으로 동질적인 아시아 인구에서 수행되었으므로, 이 E-value는 잔여 인종 혼란이 효과를 소거할 가능성이 낮음을 합리적으로 시사한다[
69].”
설명
Cochran의 Q와 I² 통계는 각 유전변인으로 추정한 인과효과의 이질성을 평가하는 데 쓰인다[
100]. 이질성이 관찰되면, 제안된 도구 중 하나 이상에서 도구변수 가정이 성립하지 않을 수 있음을 의미한다. E-value는 미측정 혼란이 결과를 설명할 수 있는 정도를 정량화한다[
69]. E-value가 크면, 특히 조상효과에 의한 혼란이 비영효과를 설명할 가능성이 낮음을 보강한다.
주요 결과에 대한 민감도 분석(항목 13a)
가정 위반에 대한 결과의 견고성을 평가하기 위해 수행한 민감도 분석을 보고한다.
예시
“고정효과 역분산가중 및 Egger 회귀 추정치는 CRP가 CAD 위험에 역의 인과효과를 시사한다(표 1). 그러나 대응하는 랜덤효과 분석은 설득력 있는 인과효과의 근거가 없음을 시사한다. 더 나아가 단순 중앙값 추정치는 반대 방향이었다. 이는 가장 강한 유전변이는 음의 인과추정치를 보였지만, 다수의 유전변이는 양의 인과추정치를 보였기 때문이다. 방법 간 불일치는 CRP의 전장유의 변이들이 모두 유효한 도구가 아님을 시사하며, 이에 근거한 인과결론은 신뢰하기 어렵다[
74].”
설명
Box 3과 7장에서 설명한 다양한 접근으로 얻은 결과를 보고ㆍ비교해, 도구변수 가정 위반에 대한 결론의 견고성을 평가해야 한다. 모든 접근의 결과가 대체로 일치하면, 효과의 존재와 크기에 대한 결론에 더 큰 확신을 가질 수 있다.
기타 분석(항목 13b)
독립 재현, 도구 검증, 시뮬레이션 등 수행한 추가 분석의 결과를 보고한다.
예시—독립 재현
“유전적으로 예측된 체질량지수와 허리둘레의 ‘흡연자일 위험’과의 관련성은 TAG 데이터에서 재현되었다(각각 1.19 [1.06–1.33], 1.32 [1.15–1.52]) [
51].”
예시—도구 검증
“MR-PRESSO는 심부전에서 1개, 관상동맥질환에서 6개, 동맥성 고혈압에서 11개의 이상치(outlier) SNP를 확인하였다. 이상치 보정은 심부전(1.13; 95% CI, 1.08–1.17), 관상동맥질환(1.08; 95% CI, 1.06–1.10), 동맥성 고혈압(1.10; 95% CI, 1.08–1.12)의 OR 추정치를 실질적으로 변화시키지 않았다. 다른 결과에 대한 MR-PRESSO 분석에서는 이상치 SNP가 확인되지 않았다[
101].”
예시—시뮬레이션
“
그림 2는 X와 Y에 대해 양의 교형질 특성 짝짓기(cross-trait assortative mating)가 있을 때 two-stage least squares regression methods (TSLS)가 양의 편향을 보임을 보여준다. 편향은 짝짓기 정도가 커질수록 비례해 증가하였다. 반면, TSLS (2)(부모 대립유전자 점수 보정)와 TSLS (3)(개인ㆍ부모 효과를 공동 모형화하고 비전달[parental non-transmitted] 대립유전자 점수를 부모 표현형의 도구로 사용)은 편향이 없었고, 위양성률은 약 5%였다[
62].”
설명
독립 재현, 도구 검증, 시뮬레이션 연구 등 수행한 민감도ㆍ추가 분석결과는 항목 8에서 설명한 바에 따라 보고해야 한다.
인과 방향성(항목 13c)
인과 방향성 평가(예: 양방향 MR)를 보고한다.
예시
“BMI 대립유전자 점수는 25(OH)D 농도와도 관련되었다(점수 1단위 증가당 −0.06% [−0.10% to −0.02%], P=0.004). 반면, 비타민 D 합성 또는 대사 대립유전자 점수는 BMI와 관련성을 보이지 않았다(합성 점수: 대립유전자 1단위당 0.01% [−0.17% to 0.20%], P=0.88; 대사 점수: 0.17% [−0.02% to 0.35%], P=0.08) [
40].”
설명
양방향 MR은 노출과 결과 각각에 관련된 독립 유전변이 집합을 사용해 양방향의 인과성을 평가하는 방법이다[
102].
비-MR 분석과의 비교(항목 13d)
관련될 때, 비-MR 분석의 추정치와 비교ㆍ보고한다.
예시
“Durbin-Wu-Hausman 내생성(endogeneity) 검정에서, 교육시간 대립유전자 점수를 이용한 도구변수 추정치는 관찰연구의 점추정치와 차이가 있다는 약한 근거가 있었다(P=0.06). 도구변수 추정치는 더 큰 음의 관련성을 시사하였다[
87].”
설명
MR 추정치와 비-MR 추정치 사이의 중요한 차이를 기술해야 한다. 연구설계마다 편향의 종류와 통계적 검정력이 다르다. MR 결과를 기존 연구 맥락과 함께 제시하면, 독자들이 MR의 장단점이 기존 증거를 지지하거나 반박하는 결과를 도출할 수 있는지 이해하는 데 도움이 될 것이다. 일반적으로 인과추론은 삼각측량(triangulation) 틀에서, 여러 접근의 근거를 종합해 제시할 수 있다[
103,
104].
결과의 추가 시각화(항목 13e)
추가 도표(예: leave-one-out 분석)를 고려한다.
예시
“Leave-one-out 분석: 각 행은 Locke 등에서 이용 가능한 전장유의 SNP 중 y축에 표시한 SNP 1개를 제외하고 수행한 ‘BMI→주관적 웰빙’ 두 표본 MR 분석을 나타낸다. 점은 해당 SNP를 제외했을 때의 효과크기, 선은 표준오차를 나타낸다. MR-Base를 사용해 특정 SNP가 BMI–웰빙 관련성을 주도하는지 확인하였다… 가장 큰 공헌을 보인 SNP는 염색체 16, fat mass and obesity associated (FTO) 유전자의 두 번째 인트론에 위치한 rs1421085였다. FTO는 여러 인구에서 비만과 반복적으로 관련되었으나, 인트론 SNP의 생물학적 결과는 아직 불확실하다. 현재로서는 시상하부에서 FTO 발현을 조절하는 역할을 하는 것으로 여겨진다. FTO의 큰 효과크기와 견고한 관련성은, 이 유전자가 다면발현 때문이 아니라 BMI 효과크기 때문에 두 표본 MR에서 가장 큰 영향을 주었음을 시사한다[
39].” (
Fig. 4,
Supplement 2).
설명
추가 도표는 결과를 시각화하고, 가정 위반을 평가하며, 영향력이 큰 점이나 이상치를 탐지하는 데에도 도움이 된다. 여기에 한 개 제외법 플롯(leave-one-out plot) [
84], 방사형플롯 (radial plot) [
105], 그리고 각 유전변이에 대해 스튜던타이즈 잔차(studentized residuals)나 Cook 거리 플롯(Cook’s distance for outlier assessment) [
32]이 포함된다.
고찰(Discussion)
고찰은 연구 해석과 타당성에 관한 핵심 이슈를 다루어야 한다[
106]. 구조화된 고찰은 저자가 결과를 과도하게 해석하는 것을 피하게 하고, 독자에게 안내 역할을 할 수 있다[
107,
108].
연구목적과 연계하여 핵심 결과를 요약한다.
예시
“약 45만 명의 포괄적 유전 자료를 바탕으로, 이 연구는 체질량지수와 체지방 분포의 차이가 흡연 행동의 여러 측면(흡연 시작 위험, 흡연 강도, 금연)에 인과적으로 영향을 준다는 근거를 제시한다. 이 결과는 비만이 흡연 시작과 금연에 미치는 역할을 부각하며, 관련 위험요인의 중요성을 낮추려는 공중보건 중재에 시사점을 제공한다[
51].”
설명
고찰은 주요 결과 요약과 그 중요성에 대한 진술로 시작하는 것이 바람직하다. 이 부분은 연구질문과 1차 결과를 상기시켜 주고, 이어지는 해석이 결과와 일관적인지 독자가 평가하게 돕는다. 요약은 주요 연구목적의 관점에서 작성하고, 사전에 설정한 가설에 초점을 맞추며, 해당 모집단에서 조사한 인과 관련성의 추정치를 보고하는 것이 바람직하다[
107].
도구변수 가정의 타당성, 기타 잠재적 편향원, 불정확성을 고려하여 연구의 한계를 논의한다. 잠재적 편향의 방향과 크기, 이를 완화하려 한 노력도 함께 기술한다.
예시
“모든 MR 분석과 마찬가지로, 이 연구도 유전도구가 관심 위험요인과 관련되고, 잠재 혼란요인과 독립이며, 대체경로(다면발현)를 통하지 않고 위험요인을 통해서만 결과에 영향을 준다는 가정을 전제하였다. 첫 번째 가정은 각 비만지표에 대해 가장 큰 GWAS에서 강건하게 관련된 변이를 사용함으로써 충족되었다. 나머지 두 가정의 충족 여부는 직접 검증하기 어렵지만, 광범위한 민감도 분석에서 뚜렷한 위반의 근거는 없었다. 둘째, 사회인구학적 요인에 의한 모집단 층화가 잠재 혼란요인이 될 수 있었다. 실제로 이전 연구에서 BMI 도구는 여성에서 사회계층 관련 요인(가계소득 낮음, 박탈감 높음 등)과 관련되었다. 그러나 남성에서는 그러한 관련성이 관찰되지 않았다. 우리 연구에서는 비만도구와 ‘흡연 시작’ 및 ‘흡연 강도’의 관련성이 남녀 각각에서, 그리고 사회적 박탈감과 연결되었을 가능성이 있는 SNP를 제외했을 때에도 일관되게 관찰되었다. 따라서 여성에서만 관찰된 ‘체지방률–금연’ 역관련성을 제외하면, 사회인구학적 요인에 의한 층화가 이러한 결과를 설명할 가능성은 낮아 보인다(추가 예시는
Supplement 2 참조) [
51].”
설명
저자는 경험적으로 완전히 검증하기 어려운 가정이 많다는 점을 고려하여, 모든 도구변수 가정의 개연성을 다루어야 한다. 예를 들어, (잔차) 유전체–표현형 혼란(모집단 구조, genetic nurture, 짝짓기 등)이 독립성 가정을 위반할 가능성을 논의할 수 있다. 위반 가능성을 평가할 때는 결과에 영향을 줄 수 있는 위반의 근원을 식별하고, 그 상대적 중요성, 유발 가능한 편향의 방향과 크기를 논의해야 한다.
또한 결과의 정밀도도 논의해야 한다. 부정확성은 연구설계의 여러 요소에서 기인할 수 있다. 예컨대 다수 SNP의 메타분석으로 얻은 도구변수 추정은 단일 SNP에 비해 보통 더 정밀하다. SNP를 발견 GWAS의 P값 기준으로 선택하였다면, 표본크기, 측정오차 등 그 GWAS의 검정력에 영향을 주는 요인을 고려해야 한다. 더 큰 데이터에서 추정할수록 SNP 효과 추정의 표준오차가 작아지므로 도구변수 추정의 정밀도는 높아진다.
해석(항목 16)
의미(항목 16a)
한계와 타 연구와의 비교를 고려하여, 결과를 신중하게 종합 해석한다.
예시
“본 멘델 무작위화 분석은 CETP (cholesteryl ester transfer protein) 억제가 심혈관 사건 위험에 미치는 인과효과가 LDL-C 또는 HDL-C 수준의 변화보다는 apoB 함유 지질단백 농도의 변화에 의해 결정될 가능성을 시사한다[
109].”
설명
전체 결과를 신중하게 해석한다. 타 연구와 비교할 때는 추정치의 차이가 왜 발생하는지 가능한 이유(도구변수 가정 위반, 불정확성, 추정방법 차이, 연구집단 차이 등)를 논의한다. 전반적 결과는 같은 질문을 다른 연구설계로 다룬 선행연구들과의 비교 맥락에서 해석해야 한다. 이렇게 하면 결과를 삼각검증(triangulation)해 해석의 신뢰도를 높일 수 있다(항목 13d 참조). 효과크기를 해석할 때는 외삽의 전제와 그것이 결과에 미친 영향을 논의한다.
기전(항목 16b)
노출과 결과 간 잠재적 인과 관련성을 매개할 수 있는 생물학적 기전을 논의하고, 유전자–환경 등가성 가정의 개연성을 검토한다. 도구변수 추정치는 특정 가정 하에서만 인과효과를 줄 수 있음을 명확히 하여 인과 언어를 신중히 사용한다.
예시
“사춘기 시기와 체중상태의 관련성은 복잡하고 양방향일 가능성이 있다. 소아의 체지방 증가는 사춘기 성숙의 조기화를 유발할 수 있으며, 남아에서는 비선형일 수 있다. 또한 여러 연구가 이른 사춘기와 이후 비만 사이의 관련성 근거를 보고하였다. 따라서 우리는 유전적으로 예측된 성인ㆍ소아 BMI를 모두 보정하려고 했고, 사춘기 시기와 다발성경화증(MS) 위험의 관련성에서 유사한 정도의 약화 현상을 관찰하였다. 다만 소아와 성인 BMI 간 강한 관련성 때문에 연령별 효과 탐색에는 한계가 있다. 그럼에도 소아 비만보다는 사춘기 이후 비만이 대사증후군(MS) 발병 위험과 가장 명확한 관련성을 보이며, 이는 사춘기 시기와 성인 비만 사이의 관련성이 사춘기 연령이 MS 위험에 미치는 효과의 가장 유력한 매개임을 시사한다. BMI와 사춘기 시기가 동일한 인과적 생물학적 경로에 속하는 것으로 보이기 때문에, 선별된 유전 변이체들이 두 노출 요인과 모두 관련되는 현상은 공유된 생물학적 기반에 기인한 수직적 다면발현(vertical pleiotropy)의 사례를 나타내며, 따라서 MR 추정값에 편향을 일으키지 않는다[
35].”
설명
유전변이를 도구로 사용될 수 있게 하는 생물학적 기전은 종종 불명확하지만, 가능한 메커니즘을 고려해 제시하는 것이 바람직하다. 이렇게 하면 MR 결과를 생물학적 기전의 맥락에서 해석할 수 있어, 인과 관련성에 대한 이해를 높일 수 있다.
MR 추정치를 인과적으로 단순 해석하기 어려운 흔한 이유는 유전자–환경 등가성 가정(
Table 2)의 타당성이 불명확하기 때문이다. 이 가정은 인구 내 유전적으로 정의된 하위집단 간 노출 차이가 중재로 인해 발생한 노출 차이와 동등하다는 것, 즉 일관성 가정(consistency assumption)의 MR 형태를 의미한다[
110]. 유전변이의 효과는 수정 시점부터 복잡하고 가변적일 수 있고, 성장ㆍ발달의 다양한 요소에 영향을 미칠 수 있다. 반면, 유전관련성 연구는 흔히 단일 시점의 표현형과의 관련성으로 변이를 확인한다. 유전변이의 일생효과는, 대개 생애 후반 특정 시점에 경험되는 환경 영향과 다를 수 있다. 이러한 생애경로 차이는 MR 추정의 유용성을 약화시키지 않는다. 예를 들어, 서로 다른 도구를 사용해 소아기와 성인기의 BMI가 다양한 건강결과에 미치는 효과를 구분할 수 있다[
111]. 반면, 저밀도 지단백(LDL) 콜레스테롤 수치 저하의 장기적 효과는 생애 초기부터 MR을 통해 추정할 수 있으며, 무작위 대조 시험에서 도출된 효과 추정치의 두 배에 달한다. 이러한 차이는 MR이 평생효과를 추정하는 반면, 임상시험은 콜레스테롤 저하 무작위 임상시험에서 불과 몇 년에 불과한 무작위 배정 기간에 대한 효과 추정치만을 제공하기 때문일 것이다. 평생 차이와 단기 콜레스테롤 저하 관련 추정치 간에 관찰되는 이러한 차이는 지질이 동맥경화성 관상동맥질환에 미치는 알려진 누적효과로부터 예상한다[
112]. 이러한 측면에서 LDL 콜레스테롤 저하를 위한 약제와 일치하도록 구성된 다양한 비중복 도구 집합들에서도 유사한 추정값이 관찰된다는 사실은 해석을 더 신뢰할 수 있도록 한다[
112]. 도구효과의 시간의존성은 MR 해석에서 중요한 이슈이며, 유전자–환경 등가성의 맥락에서 고려하는 것이 바람직하다.
임상적 관련성(항목 16c)
결과가 임상 또는 공공정책에 어떤 의미를 가질 수 있는지, 그리고 잠재적 중재의 효과크기 추정에 어느 정도 기여하는지 논의한다.
예시
“162개 SNP 각각의 정확한 기능, 심장 관련 형질과의 다면발현 정도, 그리고 이 유전변이가 심장보호 효과를 나타내는 기전에는 여전히 불확실성이 남아 있지만, 다음과 같은 결론은 가능하다… 중재는 예기치 않은 부작용에 대한 면밀한 모니터링과 함께 이루어져야 하며, 특히 장기 교육환경에 억지로 놓였을 때 적응하지 못해 건강불평등을 악화시킬 수 있는 사람들에게서 그러하다[
113].”
설명
무작위배정시험으로 검증하기 어려운 중재가 많기 때문에, MR 근거는 노출이 결과에 미치는 인과효과를 더 잘 이해하는 데 기여할 수 있다(Box 4). 다만 이러한 진술은 다른 관찰ㆍ실험 근거를 함께 고려하여 신중히 제시해야 한다. 임상ㆍ정책적 중재의 효과크기는 MR 연구에 사용된 유전변이의 효과와 다를 수 있으므로, 근거의 외삽은 명확하고 신중해야 한다.
Box 4. 멘델 무작위화의 가정 평가와 민감도 분석
인과 추정치를 해석할 때는 여러 가지를 고려해야 한다. 균질성(homogeneity) 가정이 다른 가정들(Box 2)과 함께 개연적이라면, 인과 추정치는 연구된 집단에서 노출이 결과에 미치는 평균 인과효과를 나타낸다. 균질성 가정을 둘 수 없지만 단조성(monotonicity) 가정이 개연적이라면, 인과 추정치는 국소 평균 처치효과(local average treatment effect)를 나타내는 데 사용할 수 있다[
86].
이진 노출(binary exposure)에 대한 효과 추정치를 해석할 때는 특히 주의가 필요하다[
114]. 이 경우에는 균질성과 단조성 가정이 성립할 가능성이 낮다. 또한 노출이 연속형 위험요인을 이분화한 것이라면, 배제 제한(exclusion restriction) 가정 위반의 추가 위험을 초래한다[
114].
두 집단 MR 환경에서 특히 중요한 또 하나의 고려사항은, 인과효과가 정말로 그 이진 노출에 귀속될 수 있는지 여부이다. 예를 들어, 관심 노출을 실제로 경험한 참가자가 매우 적은 노출 표본에서 두 표본 MR이 수행된 경우, 그 효과를 노출 자체의 효과로 해석하는 것은 오해를 불러일으킬 수 있다. 이때 인과 추정치는 노출에 대한 유전적 성향(genetic liability)의 효과를 반영하는 것으로 해석하는 것이 타당하다[
115,
116].
마지막으로, 해석의 중요한 요소는 시간 구간을 명확히 하는 일이다[
60]. MR 연구는 대개 노출의 ‘평생 효과(lifetime effect)’로 해석되지만, 임신부에서의 MR처럼 산전 노출을 다루는 일부 연구 설정에서는 특정 기간효과(period effect)를 연구하는 것이 더 적절할 수 있다.
일반화 가능성(항목 17)
연구결과의 일반화 가능성을 (a) 다른 인구집단, (b) 다른 노출 기간이나 시점, (c) 다른 노출 강도로 논의한다.
예시
“우리의 멘델 무작위화 연구는 비타민 D 수치와 골절 위험 사이의 선형 관계를 검토하였다. 우리는 문턱값 의존적 관계, 즉 매우 낮은 수준의 비타민 D에서만 존재할 수 있는 효과 가능성은 시험하지 않았다… 마지막으로, 비타민 D가 골절 위험 증가와 관련될 수 있다는 비유의적 경향은 건강한 사람의 선택에 기인할 수 있다(즉 매우 낮은 비타민 D 수준과 골절을 가진 참가자, 고령ㆍ허약ㆍ신체기능 저하 참가자가 GWAS 메타분석에 포함된 연구들에서 과소대표되었을 수 있다). 따라서 이 연구의 비타민 D 추정치는 이러한 고령 집단에 일반화할 수 없다[
41].”
설명
연구의 일반화 가능성은 연구가 수행된 상황과 다른 상황에 결과가 어느 정도 적용될 수 있는지를 의미한다[
117]. 예를 들어, 과거에 특정 연령대 코호트에서 수집한 결과가 현재 동일 연령대의 사람들에게 적용되지 않을 수 있다[
118].
MR 연구는 다른 방식으로도 일반화에 실패할 수 있다. 유전변이의 효과가 전 생애에 걸쳐 일정하지 않을 수 있으므로, 연구에서 산출된 효과 추정이 다른 노출 시기로 일반화될 수 있는지 고려해야 한다. 예컨대 노출의 결과에 대한 효과가 시간의존적이거나 특정 결정적 시기에만 나타난다면, 해당 기간을 벗어난 중재에 MR 추정을 적용하는 것은 오해를 낳을 수 있다. 반대로, 노출의 결과에 대한 효과가 수년에 걸쳐 누적된다면, MR은 단기 중재에 비해 효과를 과대추정할 수 있다[
60,
119].
또한 MR 추정은 대립유전자 차이에 의해 유발되는 노출범위에서만 직접 계산된다. 따라서 MR 결과의 적용은 더 넓은 노출범위에는 일반화되지 않을 수 있다. 더 나아가 MR 추정이 특정 인구 하위집단에서 도출된 것이라면, 그 범위를 넘어 일반화하기 어려울 수 있다.
기타 정보
연구비(항목 18)
이 연구와(해당되는 경우) 이 연구의 근간이 되는 데이터베이스 및 원(기) 연구의 연구비 출처와 연구비 제공자의 역할을 기술한다.
예시
“연구비: 유방암 전장유전체관련성 분석은 캐나다 정부(Genome Canada 및 Canadian Institutes of Health Research), 퀘벡주 경제ㆍ과학ㆍ혁신부(Genome Québec 및 PSR-SIIRI-701 과제), 미국 국립보건원(U19 CA148065, X01HG007492), Cancer Research UK (C1287/A10118, C1287/A16563, C1287/A10710), 유럽연합(HEALTH-F2-2009-223175, H2020 633784, 634935)의 지원을 받았다. 모든 연구와 연구비 제공자는 Michailidou 등에서 목록화되어 있다[
25]. RCR, ELA, BMB, CLR, RMM, MM, DAL, GDS는 브리스톨대학교 MRC 통합역학연구소 소속으로, Medical Research Council의 지원(과제번호 MM_UU_00011/1, MC_UU_00011/2, MC_UU_00011/5, MC_UU_00011/6, MC_UU_00011/7)을 받는다. RCR은 브리스톨대학교 de Pass VC 연구 펠로우이다. 이 연구는 브리스톨 대학병원 NHS 재단 신탁과 브리스톨대학교 NIHR 생의학연구센터의 지원을 받았다. 이 출판물의 견해는 저자 개인의 것이며, National Health Service, National Institute for Health Research, Department of Health and Social Care의 공식 입장과 일치하지 않을 수 있다. 이 연구는 Cancer Research UK(과제 C18281/A19169)와 Economic and Social Research Council (과제 ES/N000498/1)의 지원도 받았다. SEJ는 Medical Research Council(과제 MR/M005070/1)의 지원을 받는다. TMF는 유럽연구위원회(과제 323195: GLUCOSEGENES-FP7-IDEAS-ERC)의 지원을 받는다. MNW는 Wellcome Trust Institutional Strategic Support Award(과제 WT097835MF)의 지원을 받는다[
120].”
설명
연구비 출처는 연구의 설계ㆍ수행ㆍ해석에서 편향 또는 편향 인식을 유발할 수 있다[
121,
122]. 특히 연구비 제공자가 자사의 상업적ㆍ학문적ㆍ기타 이해관계에 유리한 결과에 이해관계를 가지는 경우 그러하다[
123]. 저자는 모든 연구비 출처를 공개하고, 연구질문 개발, 자료 수집ㆍ분석, 연구자 선정, 결과 검토, 원고 작성, 투고 승인 등에서 연구비 제공자의 역할을 상세히 기술해야 한다. 고용주, 정치 임명자, 정부 연구자 등의 영향도 있을 수 있다. 연구비 출처를 기술하면 독자가 연구비 제공자의 잠재적 영향하에서 작업의 신뢰성을 평가할 수 있다. 바이오뱅크 등 저장소ㆍ데이터베이스의 연구비 출처도 공개해야 하며, 이들 기관 역시 연구의 무결성에 영향을 미칠 수 있는 상업적 이해를 가질 수 있다[
124,
125].
모든 분석에 사용한 데이터를 제공하거나, 데이터 접근 위치와 방법을 논문에 명시하고, 해당 출처를 참고문헌에 제시한다. 재현을 위한 통계코드를 제공하거나, 공개 접근이 가능한지 여부와 위치를 보고한다.
예시
“데이터 공유: 이 논문에 보고된 데이터는 UK Biobank에 직접 신청하여 이용할 수 있다. UK Biobank 및 CARDIoGRAMplusC4D 컨소시엄에서의 결과와의 유전관련성은 보충자료에 제공되어 있다. 주성분분석을 포함한 멘델 무작위화 분석 구현을 위한 R 소프트웨어 코드는 보충노트에 제공되어 있다[
126].”
설명
원자료는 분석을 평가하거나 복제하려는 독자ㆍ연구자에게 필요하다. 많은 연구비 제공자와 학술지는 데이터 공유를 장려ㆍ요구하며, 저자가 제출해야 할 데이터 공유 성명 내용에 대한 지침을 제공한다. 데이터 공유는 “연구과정의 불가분의 일부”라는 합의가 형성되고 있다[
127]. 이상적으로는 연구기획 단계에서 데이터 공유계획을 수립하여 연구계획서와 논문에 기술해야 한다. 최소한 어떤 데이터가 이용 가능한지(개별 참가자 자료, 통계분석계획, 연구 관련 문서, 바이오뱅크 또는 데이터베이스 정보 등), 데이터 접근방법, 데이터 보유 주체의 연락처, 공유 메커니즘을 제시해야 한다. 또한 데이터 이용 가능 기간 제한, 요청 심사절차ㆍ기준(예: 연구계획 요구, 심의위원회 검토), 비용 부과 여부 등을 기술해야 한다. 데이터가 연합분석(federated analyses)으로만 이용 가능하다면 이를 명확히 밝혀야 한다. 데이터 출처가 다수이고 조건이 상이할 때는 서술문 대신 표 형식을 고려할 수 있다.
모든 저자는 잠재적 이해관계를 모두 선언해야 한다.
예시
“이해관계: 모든 저자는 ICMJE 통합 공개 양식(
www.icmje.org/coi_disclosure.pdf)을 작성하였다. ARC, DG, TT, JV, REW, GH, RM, SS, SB, GDS, MVH, IT, AD는 제출 작업에 대해 어떤 기관의 지원도 받지 않았고, 지난 3년간 제출 작업에 이해관계를 가질 수 있는 기관과의 재정적 관계가 없었으며, 제출 작업에 영향을 미칠 수 있는 기타 관계나 활동도 없다. MRM은 Pfizer의 연구비, GlaxoSmithKline의 비재정적 지원을(제출 작업 외) 보고하였다. NMD는 연구 수행 중 ESRC와 MRC의 연구비, 제출 작업 외의 관련 없는 연구에 대해 GRAND/Pfizer의 연구비를 보고하였다. AET는 제출 작업 외 Pfizer의 연구비를 보고하였다. LDH는 연구 수행 중 MRC의 연구비를 보고하였다. DW는 연구 수행 중 NIH의 연구비를 보고하였다[
89].”
설명
연구자와 상업적ㆍ기타 기관 간 재정적 연결, 강한 이념ㆍ지적 신념은 연구설계ㆍ수행ㆍ보고에서 편향을 유발할 수 있다. 이러한 이해관계를 공개하지 않으면 연구에 대한 공적 신뢰가 약화된다[
128]. ICMJE에 따르면, “이해관계는 1차 관심사(예: 환자 복지 또는 연구의 타당성)에 관한 전문가적 판단이 2차 관심사(예: 금전적 이익)에 의해 영향을 받을 수 있을 때 존재한다. 이해관계에 대한 인식은 실제 이해관계만큼 중요하다[
129].” 저자는 독자가 관련하다고 볼 수 있는 모든 사항을 공개하는 쪽으로 판단해야 한다.
결론
STROBE-MR 보고지침은 MR 연구에서 ‘무엇을 계획했고, 무엇을 수행했으며, 무엇을 발견했는지’를 명확히 소통하도록 돕는 최소 항목 집합을 제시한다. 고전적 역학 연구설계(코호트, 환자-대조군, 단면연구)에 대한 STROBE 지침과 유사하게[
25,
26], 이 지침의 목표는 연구 수행을 규정하거나 창의성을 제한하는 것이 아니라, 명료하고 포괄적인 보고를 촉진하여 연구의 질, 한계, 결과의 일반화 가능성에 대한 평가를 가능하게 하는 데 있다. 체크리스트는 MR 연구의 방법론적ㆍ보고 질을 평가하는 공식 도구가 아니며, 질 척도로 변환되어서는 안 된다[
130,
131]. STROBE-MR은 MR 연구의 설계ㆍ수행을 위한 공식 가이드라인으로 보아서는 안 된다. 다만 일부 항목과 이 E&E 문서는 특히 MR 연구 경험이 적은 연구자에게 방법론적 의사결정을 알리는 데 유용할 수 있다.
독자의 의견을 환영하며, 체크리스트ㆍ설명ㆍ예시 개선 제안을 부탁한다. 체크리스트와 E&E 문서는 전용 웹사이트(
https://www.strobe-mr.org/)에서 지속적으로 최신 상태로 유지될 것이다. 학술지는 저자 지침에 저자에게 기대하는 바를 명확한 언어로 포함하여 본 지침 채택을 권장한다. 예를 들어, 학술지는 저자에게 완성된 체크리스트 제출을 요구하고, 심사자에게 이를 심사의 일부로 활용하도록 요청할 수 있다[
28]. STROBE-MR은 보고지침과 자원을 모아 제공하는 EQUATOR Network 웹사이트(
https://www.equator-network.org/)에도 포함될 것이다[
132]. 또한 우리는 체크리스트와 E&E 문서의 타국어 번역 시도에 참여하기를 희망한다.
-
Authors’ contribution
ME, GDS, and JBR contributed equally to this manuscript. GDS, NMD, ND, ME, Valentina Gallo, RMG, JPTH, CL, EWL, JBR, RCR, VWS, SAS, NJT, Anne Tybjaerg-Hansen, TJV, BARW, and James Yarmolinsky contributed to the content and elaboration of the STROBE-MR checklist. All authors contributed to the writing of the article and approved of its final version. VWS, RCR, and BARW prepared the first draft of the checklist and discussion material for the workshop. VWS and JBR undertook the practical coordination of STROBE-MR. ME and GDS initiated STROBE-MR and organized the workshop; ME obtained the funding. ME, GDS, and JBR oversaw the project. JBR is the guarantor. The corresponding author attests that all listed authors meet authorship criteria and that no others meeting the criteria have been omitted.
-
Conflict of interest
All authors have completed the ICMJE uniform disclosure form at www.icmje.org/disclosure-of-interest/ and declare: support from the SNSF, NIHR Biomedical Research Centre at University Hospitals Bristol, Weston NHS Foundation Trust, and University of Bristol for the submitted work; no financial relationships with any organisations that might have an interest in the submitted work in the previous 3 years; EWL (head of research at The BMJ) played no part in the peer review or decision making of this paper at the editorial level, and contributed solely as an author; no other relationships or activities that could appear to have influenced the submitted work.
-
Funding
All STROBE-MR Initiative members are volunteers. Support for this initiative was provided by the International Swiss National Science Foundation (SNSF), UK National Institute for Health Research (NIHR) Biomedical Research Centre at University Hospitals Bristol, Weston NHS Foundation Trust, and the University of Bristol. The UK Medical Research Council (MRC) and University of Bristol support the MRC Integrative Epidemiology Unit (MC_UU_00011/1). RCR is a de Pass Vice Chancellor’s Research Fellow at the University of Bristol. NMD is supported by a Norwegian Research Council (grant 295989). SAS was supported by an NWO/ZonMW Veni grant (91617066). NJT is a Wellcome Trust Investigator (202802/Z/16/Z), is the principal investigator of the Avon Longitudinal Study of Parents and Children (MRC and WT 217065/Z/19/Z), is supported by the University of Bristol NIHR Biomedical Research Centre (BRC-1215-2001) and the MRC Integrative Epidemiology Unit (MC_UU_00011), and works within the Cancer Research UK Integrative Cancer Epidemiology Programme (C18281/A19169). JPTH is supported by NIHR Bristol Biomedical Research Centre at University Hospitals Bristol and Weston NHS Foundation Trust and the University of Bristol; is a member of the MRC Integrative Epidemiology Unit at the University of Bristol; and is an NIHR senior investigator (NF-SI-0617-10145). ME is supported by the Swiss National Science Foundation (grant 189498). JBR is supported by the Canadian Institutes of Health Research (365825; 409511), Lady Davis Institute of the Jewish General Hospital, Canadian Foundation for Innovation, NIH Foundation, CRUK, Genome Québec, Public Health Agency of Canada, and Fonds de Recherche Québec Santé. TJV was funded by US National Institutes of Health grant R01 CA222147. BARW is funded by the Economic and Social Research Council South West Doctoral Training Partnership 1+3 PhD Studentship Award (ES/P000630/1). Support from Calcul Québec and Compute Canada is acknowledged. TwinsUK is funded by the Welcome Trust, MRC, European Union, the NIHR funded BioResource, Clinical Research Facility and Biomedical Research Centre based at Guy’s and St Thomas’ NHS Foundation Trust in partnership with King’s College London. These funding agencies had no role in the design, implementation or interpretation of this study. The views expressed are those of the authors and not necessarily those of the SNSF, NIHR, Weston NHS Foundation Trust, or University of Bristol, NHS, MRC, or Department of Health and Social Care. Funders had no role in the design, conduct or results interpretation in this project.
-
Data availability
Not applicable.
-
Acknowledgments
We thank everyone who commented on previous versions of the STROBE-MR checklist; Jeremy Labrecque, Philip Haycock, and Ryan Au Yeung for their helpful suggestions; and the Medical Research Council Integrative Epidemiology Unit within the Bristol Medical School, University of Bristol, UK, for hosting the May 2019 workshop.
Supplementary materials
Supplement 1. STROBE-MR checklist of recommended items to address in reports of Mendelian randomization studies; skrv065837.ww1.pdf (101.5KB, pdf).
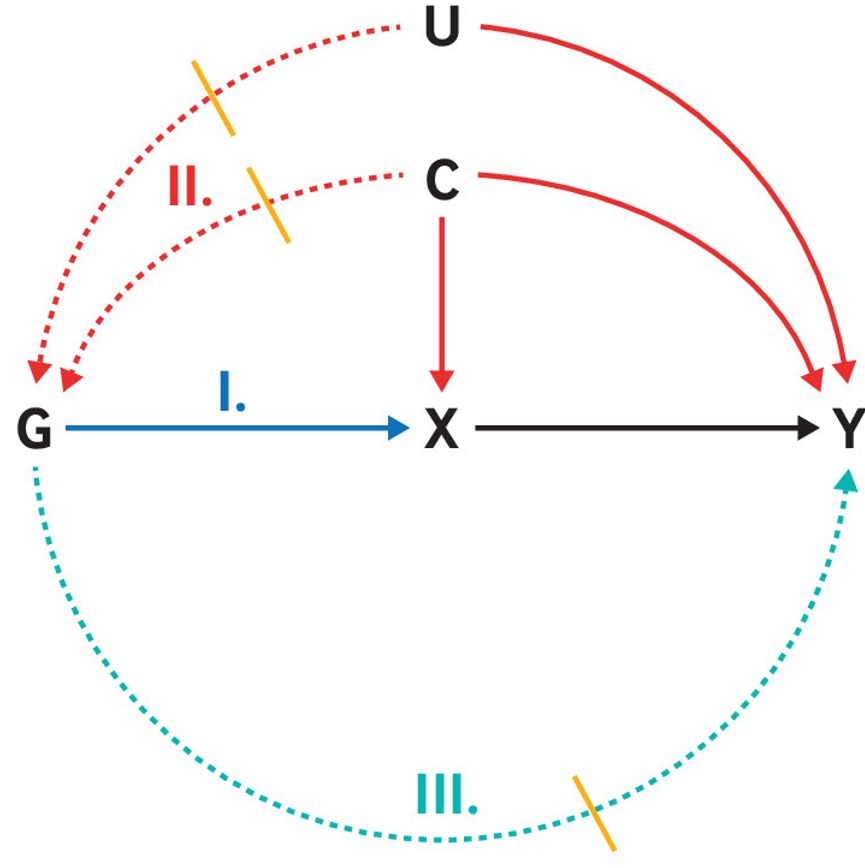
Fig. 1.도구변수(IV) 분석의 가정을 보여 주는 표준 인과도. 유전변이 G를 노출 X의 도구변수(대리변수)로 사용해, 결과 Y에 대한 X의 인과효과를 평가한다. IV 가정은 다음과 같다: I. 관련성: 유전변이 G는 관심 노출 X와 관련되어 있다. II. 독립성: 유전변이 G는 결과 Y와 공유하는 미측정 원인이 없다. III. 배제 제한: 유전변이 G는 관심 노출 X에 대한 잠재적 효과를 통한 경우를 제외하고는 결과 Y에 영향을 미치지 않는다. 실선 화살표=인과효과; 점선 화살표=IV 가정에 의해 금지되는 인과효과. 참고로, IV 가정을 만족하는 인과도는 다른 방식으로도 그릴 수 있다(예: 유전변이 G가 노출 X를 직접 원인으로 하지 않아도 된다). 반대로, 도식에 그려지지 않은 다른 경로는 IV 가정을 위반할 수 있다(예: 선택 편향은 독립성 가정을 위반할 수 있음).
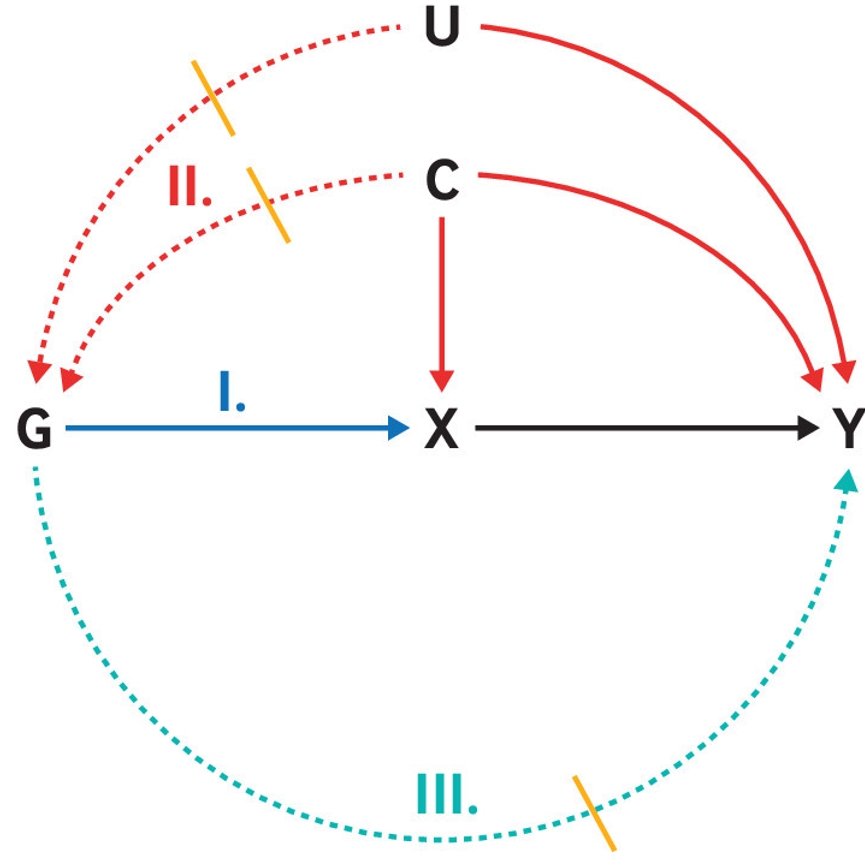
Fig. 2.멘델 무작위화 연구 유효성 검증을 통과한 UK Biobank 참가자 수. 그림은 Mountjoy 등, 2018에서 허가받아 재수록함[
87].
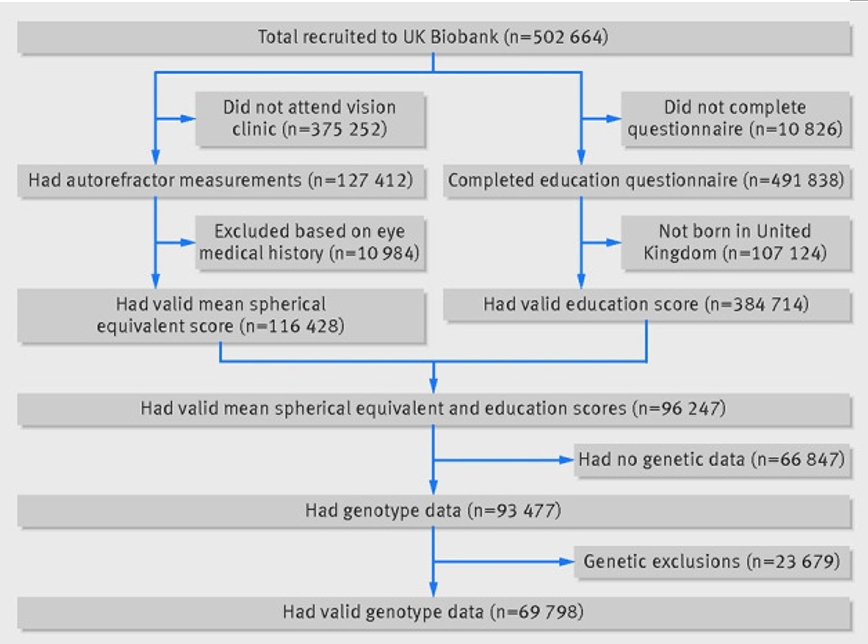
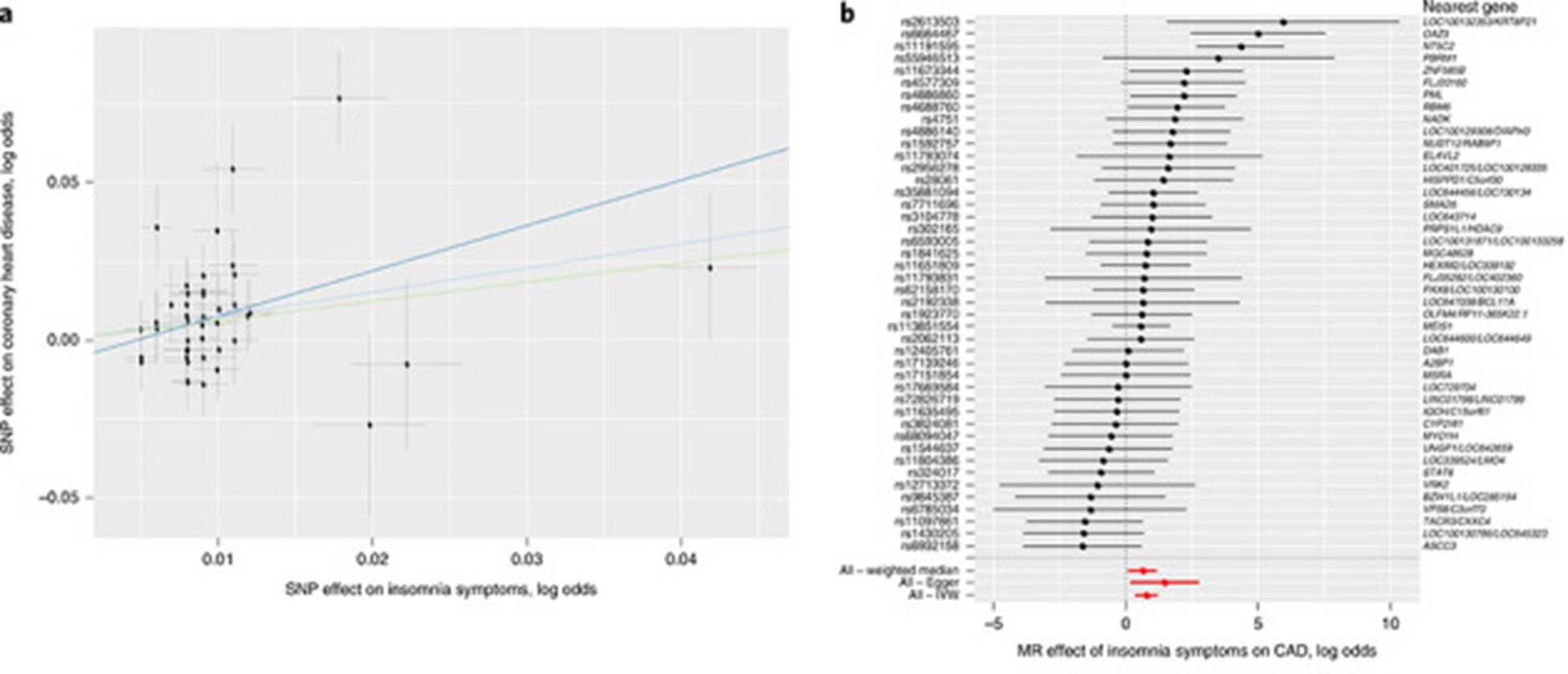
Fig. 3.“불면 증상의 인과적 관계. (A) 잦은 불면 증상과 관련된 SNP와 관상동맥질환(CAD) 간의 관련성을 제시한다. 각 대립유전자(allele)당 잦은 불면 증상 위험과의 관련성(가로축) 대비, 각 대립유전자당 CAD 위험과의 관련성(세로축)을 산점도로 표시하였으며, 각 점을 둘러싼 검은색 세로ㆍ가로 선은 각 다형성의 95% 신뢰구간(CI)을 나타낸다. 세 가지 서로 다른 MR 관련성 검정결과를 함께 제시한다. (B) 유전적으로 증가한 불면 위험이 CAD에 미치는 효과 추정치를 포레스트 플롯으로 제시한다. 가장 가까운 유전자는 플롯 오른쪽에 표시한다. 각 SNP에 대해 추정치의 95% CI(회색 선분)와 IVW MR, MR-Egger, 가중 중앙값(weighted-median) MR 결과(빨간색)를 함께 제시한다. MR 분석에 사용한 각 GWAS의 표본 크기는 다음과 같다: 잦은 불면 증상(사례 n=129,270; 대조군 n=108,352), CAD(사례 n=60,801; 대조군 n=123,504).” 도판은 Lane 등(2019)에서 허가를 받아 재인용하였다[
98].
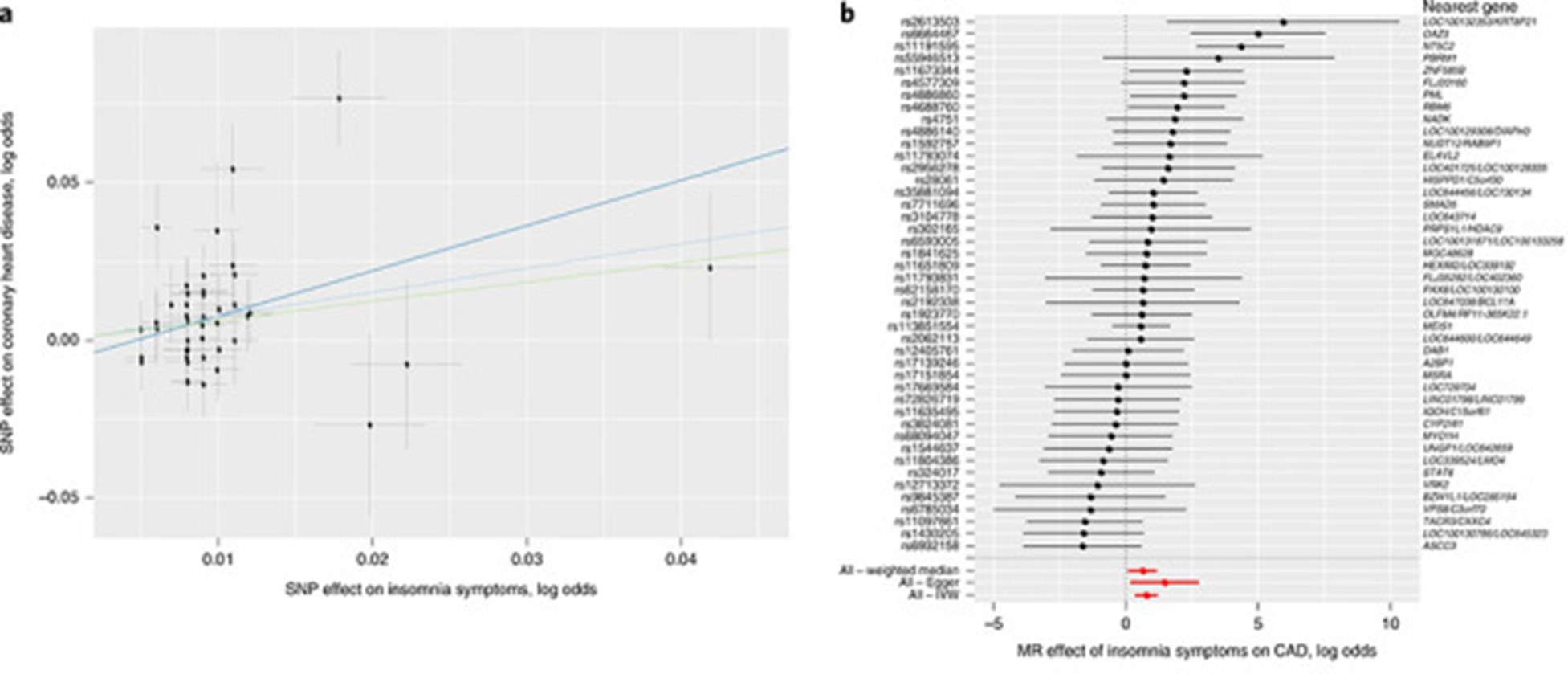
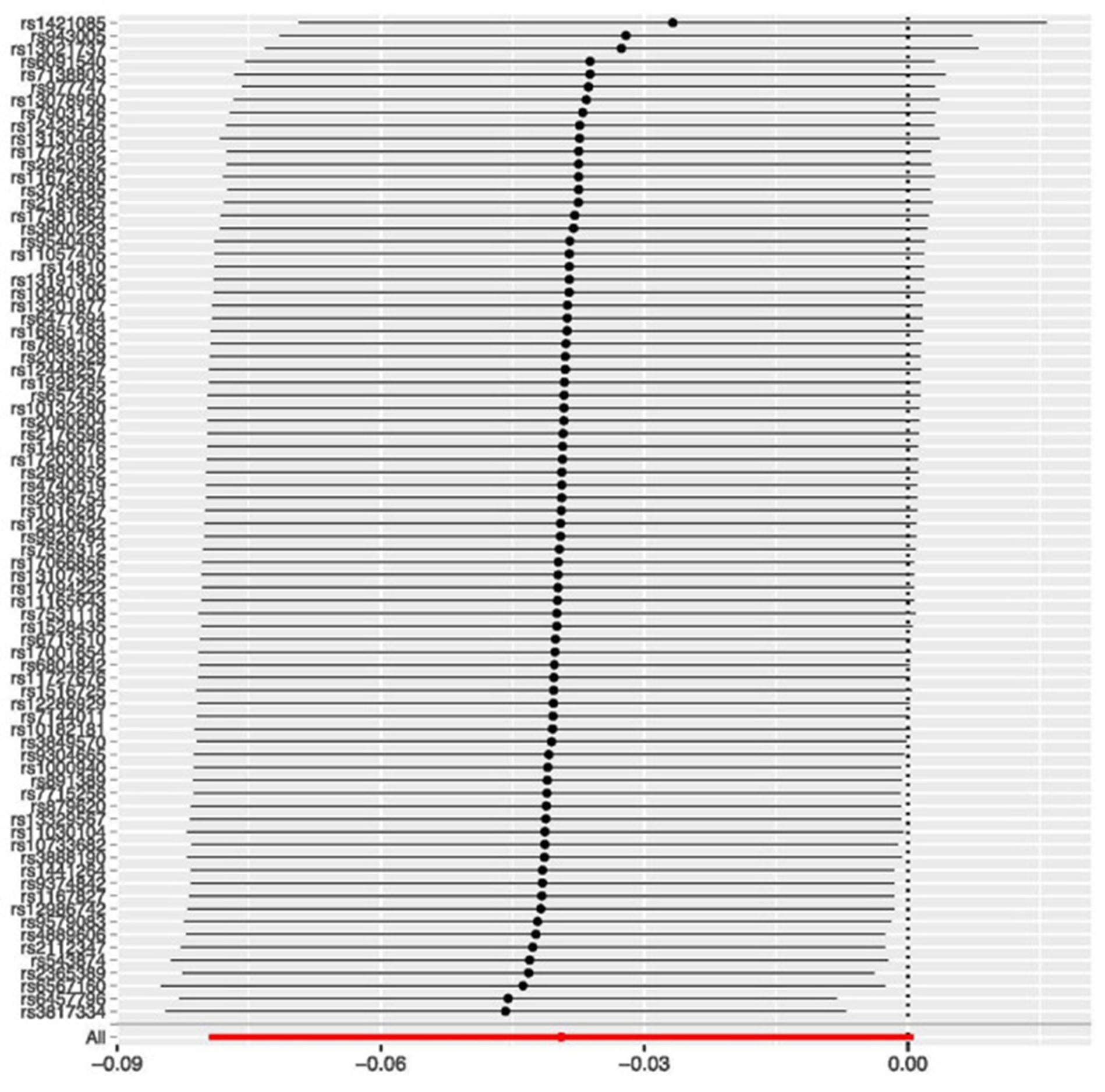
Fig. 4.Leave-one-out 분석. 도판은 Wootton 등(2018)에서 허가를 받아 재인용하였다[
39].
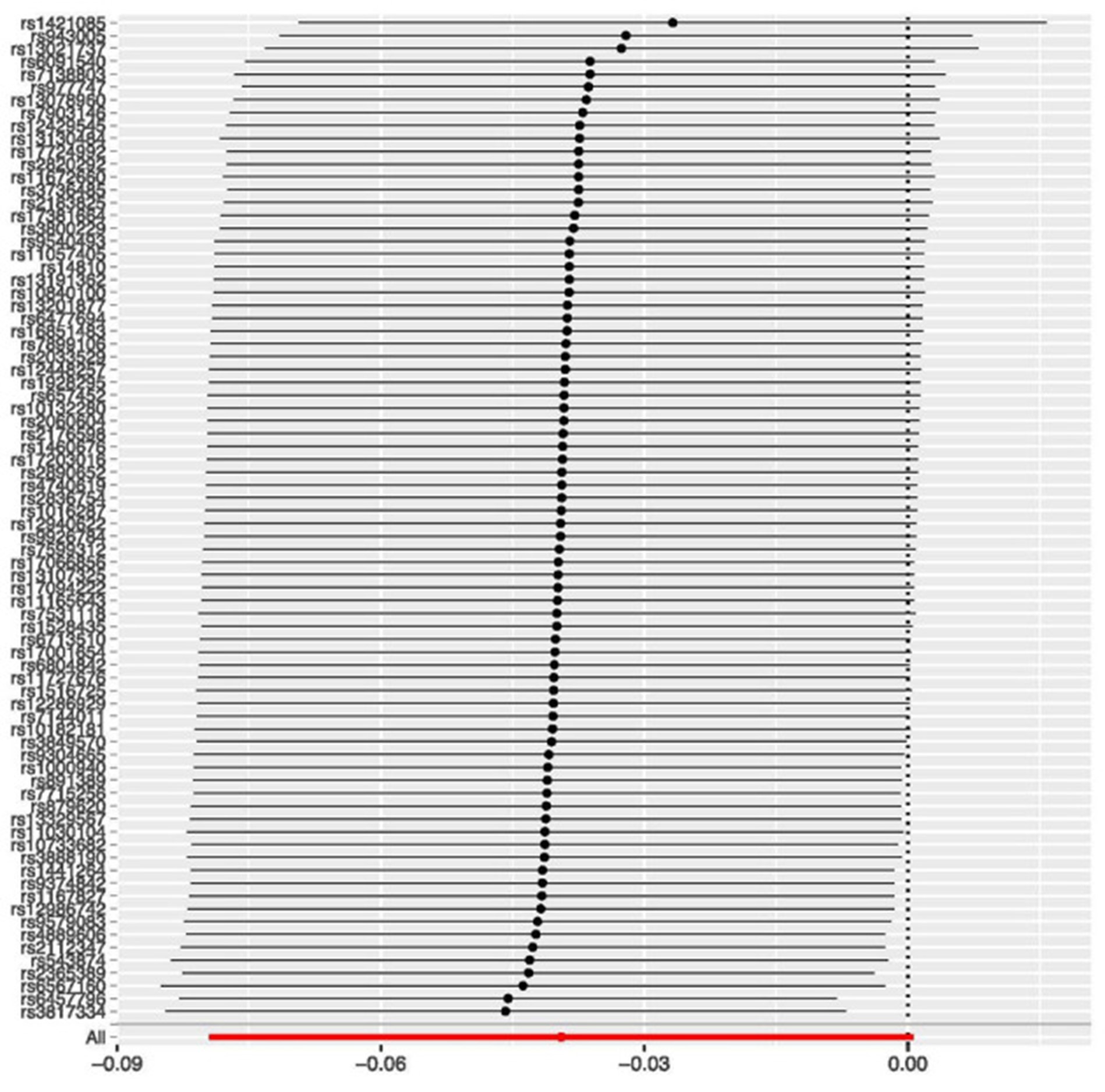
Table 1.STROBE-MR 체크리스트가 다루는(또는 다루지 않는) 연구설계
|
STROBE-MR이 다루는 연구 유형 |
STROBE-MR이 다루지 않는 연구 유형 |
|
단일 표본(집단)(one-sample) MR 연구 |
전장유전체 관련성 분석(GWAS) |
|
두 표본(집단)(two-sample) MR 연구 |
시퀀싱 연구 |
|
동일 논문에서 GWAS 이후 수행되어 함께 보고된 MR 연구 |
유전자 발현 연구 |
|
단일 또는 두 표본 MR 중 다중 노출a)ㆍ다중 결과(또는 둘 다)를 다루는 연구 |
전통적 관찰연구 |
Table 2.
|
용어 |
설명 |
|
MR |
유전 변이를 이용해 조절 가능한 노출(예: 체질량지수, 알코올 섭취, 혈중 지질단백, 교육시간, C-반응단백, 혈청 25-하이드록시 비타민 D)이 질병 위험이나 다른 결과에 미치는 인과적 관계를 추정하는 방법론적 접근이다. 대부분의 MR 연구는 유전변이를 도구변수로 사용하는 도구변수 분석의 형태로 수행된다. |
|
단일 표본(one-sample) MR |
동일한 연구 표본에서 유전 변이(SNP), 노출, 결과가 모두 측정되어, SNP 노출(G X) 및 SNP 결과 (G Y) 연관을 같은 개별 수준 자료 (individual level data) 에서 추정하는 연구 설계이다. 유전도구변수 (genetic instrumental variable)는 MR 의 3 가지 가정(①노출과 연관, ② 노출을 통한 경로만으로 결과에 영향, ③ 교란 요인과 독립 을 충족해야 한다. 단일 표본 MR은 일반적으로 2 단계 최소제곱법(two stage least squares, 2SLS) 을 통해 구현된다. 1 단계에서는 유전 변이로부터 노출을 예측하고, 2 단계에서는 예측된 노출값과 결과의 관계를 분석하여 인과효과를 추정한다. 개별 수준 자료를 이용하므로, 교란 요인과의 연관 검증, 상호작용(interaction), 비선형성(nonlinearity), 하위표본 분석(subgroupanalysis)이 가능하다. 단일 표본 MR에서는 약한 도구로 인한 편향이 전통적 회귀분석의 교란된 추정치 방향으로 치우치는 경향이 있다. |
|
두 표본(two-sample) MR |
서로 다른 독립 표본에서 G X, G Y 연관을 추정하고, 이를 메타분석 혹은 Wald ratio 방식으로 결합하는 연구 설계이다. 각 표본에서 유전 변이별 요약 통계(summary level data)가 필요하며, 개별 수준 자료는 요구되지 않는다. 이 방법은 역인과(reverse causality) 가능성을 점검하고, 인과 방향(direction of causality)을 명확히 하기 위해 활용된다. 각 변수의 유전 도구변수는 MR의 3 가지 기본 가정 관련성 · 독립성 · 배제 제한을 각각 충족해야 하며, 양 방향의 분석간 통계적 검정력(statistical 차이와 도구의 유효성 차이를 고려하여 해석해야 한다. |
|
양방향(bidirectional) MR |
한 집합의 도구변수로 ‘노출→결과’ 효과를, 다른 독립 집합으로 ‘결과→노출’ 효과를 시험해 인과 방향성을 더 잘 파악하는 접근이다. |
|
도구변수(instrumental variables) |
관심있는 노출과 통계적으로 유의미한 연관을 가지며, 교란 요인과는 독립적이고, 노출을 통한 경로 외에는 결과에 영향을 미치지 않는 변수이다. |
|
도구변수 가정(핵심 MR 가정) |
관련성(relevance: 유전변이가 관심 노출과 관련성), 독립성(independence: 유전변이가 결과와 공유하는 미측정 원인이 없음), 배제 제한(exclusion restriction: 유전변이가 관심 노출의 효과 경로 외에는 결과에 영향 없음)의 가정을 포함한다. |
|
도구변수 가정 평가 |
가정의 개연성은 여러 가지 방법으로 점검할 수 있다. 관련성은 유전 변이와 관심 노출 간의 연관을 검증하고, 배제 제한 가정은 다면발현이나 교란 요인과의 관계를 검정하여 정황 증거를 제시할 수 있다. 배제 제한 가정은 위반 여부를 직접적으로 검증할 수 없지만, MR Egger 회귀, weighted median 방법 등으로 위반 가능성을 평가할 수 있다. |
|
유전자–환경 등가성(gene–environment equivalence) |
유전변이에 의해 유도된 노출 차이가 환경 요인에 의해 유도된 노출 차이와 건강 결과에 미치는 하위 효과가 동일하다는 개념이다. 이 가정이 충족되어야 MR 추정치를 정책적 개입 효과로 해석 가능하다. |
|
유전변이(genetic variant) |
한 집단 내에서 관찰되는 DNA 서열의 변이를 말한다. 전형적으로 단일염기다형성(SNP)을 가리킨다. |
|
단일염기다형성(SNP) |
DNA의 단일 염기가 의미 있는 빈도로 집단 내에서 변이를 보이는 유전변이다. 보통 두 개의 대립유전자(예: A, C, G, T)를 가지며, 형질과 관련된 경우 하나는 형질 증가, 다른 하나는 감소와 관련된다. MR에서는 조절 가능한 노출의 도구변수로 가장 흔히 사용된다. |
|
가닥 정렬(strand alignment) |
노출 GWAS와 결과 GWAS에서 보고된 대립유전자가 동일한 DNA 가닥 기준으로 일치하도록 맞추는 절차이다. 팔린드롬(palindromic) SNP(G/C, A/T)는 두 가닥에서 동일하게 보여 혼동을 유발할 수 있어 주의가 필요하다. 가닥을 일치시키지 않으면 효과대립유전자 식별의 모호성이 생긴다. MR 또는 메타분석 수행 시, 노출 GWAS 와 결과 GWAS 에서 보고된 대립유전자가 동일한 DNA 가닥 기준으로 일치하도록 맞추는 절차이다. |
|
대립유전자 점수(allele score) |
여러 SNP의 정보를 결합해 하나의 변수로 만든 것으로, 특정 형질(예: 혈압)과 관련된 SNP들을 합성해 MR에서 노출을 예측하는 데 쓴다. 유전위험점수(genetic risk score), 다유전자 점수(polygenic score), 유전 예측점수라고도 한다. |
|
관련성불평형(LD) |
두 개 이상의 좌위에서 대립유전자가 무작위가 아닌 방식으로 공동 분포하는 현상이다. 일반 인구에서 보통 좁은 유전체 구간 내에서 관찰되며, MR에서 편향의 잠재 원인이 될 수 있다. |
|
r² |
두 유전 좌위 간 LD의 상관을 정량화하는 지표이다(r²=1은 완전 상관). 노출 변이의 분산 설명력을 나타내는 R²(도구 강도 계산에 쓰임)와 혼동하지 않도록 한다. LD는 보통 인접한 염색체 구간 내의 유전변이들 수 킬로베이스 수십 킬로베이스 범위 에서 관찰되며, 이는 재조합(recombination)이 세대를 거듭해도 완전히 무작위화되지 않기 때문에 발생한다. MR 연구에서는 LD가 독립적 도구변수(independent instrumental variables) 가정의 위반을 초래할 수 있다. 즉 선택된 SNP들이 서로 높은 LD에 있다면, 각각의 SNP가 동일한 인과 신호를 중복 반영하거나, 다른 유전자의 효과를 혼합(reflection)함으로써 함으로써 효과 추정치의 편향(bias) 또는 과대평가(overestimation)가 발생한다. |
|
도구 강도 검정(test of instrument strength) |
유전변이와 노출 간 관련성의 크기를 평가한다. 부분 F 통계량(partial F)이나 R²가 흔히 사용된다. |
|
차이 검정(test for difference) |
다변량 보정 관찰연구 추정치와 MR 추정치의 차이가 단순 추정오차를 넘어서는지 평가한다(예: 하우스만 검정). 일반최소제곱법(ordinar y least squares, OLS 에서 얻은 추정치와 2단계 최소제곱법(two stage least squares, 2SLS)에서 얻은 추정치의 차이를 그 분산 차이로 나누어(statistical ratio of their variance difference) 통계적으로 평가한다. 차이가 통계적으로 유의하면(P<0.05), 관찰연구 추정치가 교란(confounding)이나 역인과(reverse causality)의 영향을 받을 가능성 이 있거나, MR 의 핵심 가정(예: 배제 제한)이 위배되었을 가능성을 시사한다. |
|
수평 다면발현(horizontal pleiotropy) |
유전변이가 관심 노출과 무관한 경로로 결과에 영향을 미치는 현상이다. 배제 제한 가정 위반이며 MR 편향의 원인이다. |
|
약한 도구 편향(weak instrument bias) |
도구로 사용한 유전변이가 관심 노출과의 관련성이 약할 때 발생하는 편향이다. 단일 표본 분석에서 부분 F<10이면 약한 도구 가능성이 높다는 경험법칙이 있으나, 10을 넘어도 편향이 생길 수 있다(이는 P=0.05의 이분법과 유사한 경험적 기준일 뿐이다)[20] |
|
콜라이더 편향(collider bias) |
유전변이와 또 다른 핵심 변수(예: 결과)의 공통 효과에 대해 조건부화할 때 생길 수 있는 편향이다. 통계적 조정(변이와 결과 모두에 의해 생성된 공변량을 모형에 포함)이나 표본추출 과정(예: 입원이 변이와 다른 요인에 의해 좌우되는 병원 환자 표본 분석)에서 발생할 수 있다. |
|
승자의 저주(winner’s curse) |
가장 유의한 SNP의 효과가 실제보다 과대 추정되는 현상이다. 일반적으로 P값 문턱으로 가장 강한 관련성만 선택할 때 발생한다. |
|
데이터(data) |
참가자의 개별 수준 자료(예: 표현형·유전형) 또는 SNP 수준의 표현형–관련성 요약치(요약수준 자료)를 가리킨다. |
Table 3.멘델 무작위화(MR) 연구 보고에서 다뤄야 할 권장 항목(STROBE-MR 체크리스트)
|
항목 번호 |
구역 |
체크리스트 항목 |
|
1 |
제목과 초록 |
연구의 주요 목적이 MR일 때, 제목 및/또는 초록에 MR을 연구설계로 명시한다. |
|
서론
|
|
|
2 |
배경 |
보고하는 연구의 과학적 배경과 근거를 설명한다. 노출은 무엇인가? 노출과 결과 간 잠재적 인과 관련성은 개연적인가? MR이 연구질문을 다루는 데 유용한 이유를 제시한다. |
|
3 |
목적 |
사전에 규정된 인과 가설(있다면)을 포함하여 구체적 목적을 명확히 기술한다. MR은 특정 가정하에서 인과효과를 추정하려는 방법임을 명시한다. |
|
방법
|
|
|
4 |
연구설계와 데이터 출처 |
논문 서두에 연구설계의 핵심 요소를 제시한다. 연구의 모든 단계에 대한 데이터 출처를 나열한 표를 포함하는 것을 고려한다. 분석에 기여한 각 데이터 출처에 대해 다음을 기술한다: a) 환경(Setting): 가능하면 연구설계와 기반 모집단을 설명한다. 이용 가능한 경우 모집·노출·추적·자료수집 기간을 포함해 장소·위치·관련 날짜를 기술한다. b) 참가자: 선정기준, 선정의 원천과 방법을 제시한다. 표본 수, 본 분석 이전에 수행한 검정력/표본 수 계산 여부를 보고한다. c) 유전변이의 측정·품질관리·선정을 기술한다. d) 각 노출·결과·기타 변수의 평가방법과 질병의 진단기준을 기술한다. e) 해당 시 윤리 심의 승인과 연구참여 동의의 세부를 제시한다. |
|
5 |
가정 |
본 분석의 세 가지 핵심 도구변수 가정(관련성, 독립성, 배제 제한)과 추가/민감도 분석에 필요한 가정을 명시한다. |
|
6 |
통계방법: 본 분석 |
사용한 통계방법과 통계치를 기술한다. a) 정량 변수 처리(척도, 단위, 모형)를 기술한다. b) 유전변이 처리와 가중치 선택(해당 시)을 기술한다. 유전변이 전처리(품질 관리, LD 클럼핑, 가닥 정렬 팔린드롬 처리)와 분석 절차를 기술한다.c) MR 추정량(예: 2단계 최소제곱, Wald 비)과 관련 통계, 포함 공변량, 두 표본 MR의 경우 두 표본에서 동일 공변량 세트를 사용해 보정했는지 기술한다. 도구 강도 평가(F 통계량), 2 단계 최소제곱법 (2SLS) 또는 요약통계 기반 MR(IVW, MR Egger 등) 방법을 명시한다 d) 결측치 처리방법을 명확히 한다. e) 해당 시 다중 검정처리 방법을 기술한다. 유전 변이 노출 (G X), 유전 변이 결과 (G Y) 연관 산출법과 다중 검정 보정을 기술한다. |
|
7 |
가정의 평가 |
가정의 타당성을 평가하거나 근거를 제시하기 위해 사용한 방법 또는 사전 지식을 기술한다. |
|
8 |
민감도 분석 및 추가 분석 |
수행한 민감도/추가 분석을 기술한다(예: 방법 간 효과추정 비교, 독립 재현, 편향 분석 기법, 도구 검증, 시뮬레이션). |
|
9 |
소프트웨어 및 사전 등록 |
a) 사용한 통계 소프트웨어와 패키지(버전·설정 포함)를 명시한다. b) 연구 프로토콜과 세부가 사전 등록되었는지(언제·어디에) 기술한다. |
|
결과
|
|
|
10 |
기술 통계 |
a) 포함된 연구의 각 단계에서 개인 수와 제외 사유를 보고한다(플로우 다이어그램 사용 고려). b) 표현형 노출·결과·기타 변수의 요약통계(예: 평균, 표준편차, 비율)를 보고한다. c) 데이터 출처에 기존 메타분석이 포함되면, 연구 간 이질성 평가를 제시한다. d) 두 표본 MR의 경우: i. 노출 표본과 결과 표본에서 유전변이–노출관련성의 유사성에 대한 근거를 제시한다. ii. 두 표본에 중복 포함된 개인 수를 보고한다. |
|
11 |
주요 결과 |
a) 유전변이–노출, 유전변이–결과 관련성을 해석 가능한 척도로 보고한다. b) MR 추정치와 불확실성 지표를 해석 가능한 척도로 보고한다(예: 표준편차 1단위당 OR 또는 RR). c) 해당 시, 상대위험을 임상적으로 의미 있는 기간의 절대위험으로 변환하는 것을 고려한다. d) 결과 시각화를 위한 도표를 고려한다(예: 포레스트 플롯, 유전변이–결과 대 유전변이–노출 산점도). |
|
12 |
가정의 평가(결과) |
a) 가정 타당성 평가결과를 보고한다. b) 추가 통계(예: 유전변이 간 이질성 평가: I², Q 통계, E-value)를 보고한다. |
|
13 |
민감도 분석 및 추가 분석(결과) |
a) 가정 위반에 대한 주요 결과의 견고성을 평가하기 위해 수행한 민감도 분석을 보고한다. b) 기타 민감도/추가 분석결과를 보고한다. c) 인과 방향성 평가(예: 양방향 MR)를 보고한다. d) 관련될 때 비-MR 분석 추정치와 비교·보고한다. e) 추가 시각화(예: leave-one-out 분석)를 고려한다. |
|
고찰
|
|
|
14 |
핵심 결과 |
연구목적에 비추어 핵심 결과를 요약한다. |
|
15 |
한계 |
도구변수 가정의 타당성, 기타 잠재적 편향원, 불정확성을 고려해 연구의 한계를 논의한다. 잠재적 편향의 방향과 크기, 이를 줄이려 한 노력을 함께 기술한다. |
|
16 |
해석 |
a) 의미: 한계와 타 연구와의 비교 맥락에서 결과를 신중하게 종합 해석한다. b) 기전: 노출–결과 인과 관련성을 매개할 수 있는 생물학적 기전을 논의하고, 유전자–환경 등가성 가정의 개연성을 검토한다. 인과 언어는 신중히 사용하며, 도구변수 추정치는 특정 가정하에서만 인과효과를 제공할 수 있음을 명확히 한다. c) 임상적 관련성: 결과가 임상·정책에 갖는 함의와, 잠재적 중재의 효과크기 추정에 어느 정도 기여하는지 논의한다. |
|
17 |
일반화 가능성 |
연구결과의 일반화 가능성을 (a) 다른 인구집단, (b) 다른 노출 기간/시점, (c) 다른 노출 수준으로 논의한다. |
|
기타 정보
|
|
|
18 |
연구비 |
본 연구(및 해당 시, 기반 데이터베이스·원연구)의 연구비 출처와 연구비 제공자의 역할을 기술한다. |
|
19 |
데이터 및 데이터 공유 |
분석에 사용한 데이터를 제공하거나 접근 위치·방법을 명시하고, 논문 내에서 이를 인용한다. 결과 재현에 필요한 통계 코드를 제공하거나, 공개 접근 가능 여부와 위치를 보고한다. |
|
20 |
이해관계 |
모든 저자는 잠재적 이해관계를 모두 선언한다. |
Table 4.각 표현형에 사용된 GWAS 컨소시엄 개요(도표는 Wootton 등, 2018에서 허가 받아 재수록함[
39])
|
변수 |
제1저자(연도) |
컨소시엄 |
표본 수 |
인구집단a)
|
성별a)
|
|
주관적 웰빙 |
Okbay (2016) |
SSGAC |
298,420 |
유럽계 100% |
혼합b)
|
|
체질량지수(BMI) |
Locke (2015) |
GIANT |
339,224 |
유럽계 95% |
여성 53% |
|
허리–엉덩이둘레비(WHR) |
Shungin (2015) |
GIANT |
210,088 |
유럽계 100% |
여성 56% |
|
허리둘레 |
Shungin (2015) |
GIANT |
232,101 |
유럽계 100% |
여성 55% |
|
체지방률 |
Lu (2016) |
보고 없음 |
100,716 |
유럽계 89% |
여성 48% |
|
HDL 콜레스테롤 |
Willer (2013) |
GLGC |
92,860 |
유럽계 100% |
혼합b)
|
|
LDL 콜레스테롤 |
Willer (2013) |
GLGC |
83,198 |
유럽계 100% |
혼합b)
|
|
총 콜레스테롤 |
Willer (2013) |
GLGC |
92,260 |
유럽계 100% |
혼합b)
|
|
관상동맥질환(CAD) |
Nikpay (2015) |
CARDIoGRAMplusC4D |
사례 60,801; 대조 123,504 |
유럽계 77% |
혼합b)
|
|
심근경색(MI) |
Nikpay (2015) |
CARDIoGRAMplusC4D |
사례 43,676; 대조 128,199 |
혼합b)
|
혼합b)
|
|
이완기 혈압 |
Wain (2017) |
보고 없음 |
150,134 |
유럽계 100% |
여성 60% |
|
수축기 혈압 |
Wain (2017) |
보고 없음 |
150,134 |
유럽계 100% |
여성 60% |
Table 5.멘델 무작위화에서 가장 흔한 도구변수 가정과 가능한 평가ㆍ민감도 분석 예시
|
가정 |
가능한 평가 예시 |
|
관련성(Relevance): 유전변이는 관심 노출과 관련되어 있다 |
F 통계량을 보고한다. |
|
독립성(Independence): 유전변이는 결과와 공유하는 미측정 원인이 없다 |
가능한 교란변수가 유전변이(들)과 결과 모두와 어떤 관련성을 보이는지 보고한다; 모집단 층화를 어떻게 보정했는지(예: 주성분 보정) 기술한다; 변이–결과 관련성에 대한 미측정 교란 민감도 지표를 제시한다[59,68,69,75]. |
|
배제 제한(Exclusion restriction): 유전변이는 관심 노출을 통한 경로를 제외하고는 결과에 영향을 미치지 않는다 |
MR-Egger 회귀의 기울기 추정치, 절편 및 그 95% 신뢰구간을 보고한다; 음성 대조 결과 또는 음성 대조 집단을 이용한 결과를 함께 제시한다. |
|
균질성(Homogeneity, 2단계 최소제곱): 관심 노출의 인과효과는 결과에 대해 모든 개인에서 일정하다 |
서로 다른 측정 가능한 하위집단(예: 연령, 인종/민족, 성별, 사회경제적 지위)에 대한 도구변수 효과추정치를 보고한다; 연속형 결과의 경우, 도구 수준별 분산을 보고한다[72]. |
|
InSIDE (MR-Egger): 유전변이의 노출과의 관련성은 그 변이의 결과에 대한 직접효과와 독립적이어야 한다 |
이 가정을 요구하지 않는 다른 추정기(예: 중앙값 기반, 최빈값 기반검정)의 효과추정치도 함께 보고한다. |
Table 6.UK 바이오뱅크에서 흡연 및 성별 범주별 신체 크기 매개변수의 표본 특성
|
신체지표 |
전체(n=372,791) |
비흡연(n=203,735) |
과거 흡연(n=131,537) |
현재 흡연(n=37,519) |
여성(n=200,247) |
남성(n=172,544) |
|
체질량지수 (BMI) |
27.4 (4.8) |
27.1 (4.7) |
28.0 (4.7) |
27.0 (4.8) |
27.0 (5.1) |
27.9 (4.2) |
|
체중 (kg) |
78.3 (15.9) |
77.0 (15.6) |
80.5 (16.0) |
78.0 (16.3) |
71.5 (13.9) |
86.2 (14.3) |
|
신장 (cm) |
168.8 (9.2) |
168.3 (9.3) |
169.4 (9.1) |
169.5 (9.2) |
162.7 (6.2) |
175.9 (6.7) |
|
허리둘레 (cm) |
90.4 (13.5) |
88.8 (13.2) |
92.6 (13.6) |
91.2 (13.5) |
84.6 (12.5) |
97.1 (11.3) |
|
체지방률 (%) |
31.4 (8.5) |
31.5 (8.6) |
31.7 (8.2) |
29.9 (8.6) |
36.6 (6.9) |
25.3 (5.8) |
Table 7.UK 바이오뱅크 평생 흡연자(현재 흡연자+이전 흡연자)의 체질량지수 및 성별 범주별 흡연 매개변수의 표본 특성
|
흡연 지표 |
전체 (n=169,056) |
체질량지수(BMI) 범주 |
성별 |
|
저체중 (<18.5; n=816) |
정상 (18.5–25.0; n=49,017) |
과체중 (25.0–30.0; n=74,439) |
비만 (>30.0; n=44,784) |
여성 (n=81,091) |
남성 (n=87,965) |
|
흡연 시작 연령(세) |
17.3 (4.2) |
17.5 (4.8) |
17.6 (4.2) |
17.3 (4.2) |
17.1 (4.3) |
17.8 (4.4) |
16.9 (4.0) |
|
하루 평균 흡연 개비수—평생 흡연자(ever smokers) |
18.4 (10.1) |
16.6 (10.5) |
15.9 (8.6) |
18.2 (9.6) |
21.1 (11.5) |
16.1 (8.2) |
20.5 (11.2) |
|
하루 평균 흡연 개비수—현재 흡연자(current smokers)a)
|
15.8 (8.4) |
16.8 (11.1) |
15.0 (8.2) |
15.6 (8.1) |
17.3 (9.0) |
14.2 (7.3) |
|
Table 8.골절과 관련된 전장유의(genome-wide significant) 단일염기다형성(SNP)
|
염색체 위치(locus) |
후보 유전자(candidate gene) |
SNP |
유전자까지 거리(kb) |
EA |
EAF |
Odds ratio (95% CI), P |
골절 사례 수(no. of fracture cases) |
I² |
|
발견 단계a)
|
검증 단계a)
|
통합a)
|
|
2p16.2 |
SPTBN1 |
rs4233949 |
−23.21 |
G |
0.61 |
1.03 (1.02–1.05), 6.9×10−5
|
1.04 (1.05–1.05), 8.9×10−11
|
1.03 (1.02–1.04), 2.8×10−14
|
185,057 |
22.4 |
|
3p22.1 |
CTNNB1 |
rs430727 |
107.2 |
T |
0.45 |
1.03 (1.02–1.05), 1.0×10−4
|
1.03 (1.02–1.04), 1.1×10−8
|
1.03 (1.02–1.04), 5.0×10−12
|
185,057 |
0 |
|
6q22.33 |
RSPO3 |
rs10457487 |
0 |
C |
0.51 |
1.06 (1.05–1.08), 2.3×10−15
|
1.04 (1.03–1.05), 1.7×10−15
|
1.05 (1.04–1.06), 4.8×10−28
|
185,057 |
5 |
|
6q25.1 |
ESR1 |
rs2982570 |
0 |
C |
0.58 |
1.05 (1.04–1.07), 8.1×10−12
|
1.03 (1.02–1.04), 5.2×10−10
|
1.04 (1.03–1.05), 4.5×10−19
|
185,057 |
23 |
|
7q31.31 |
WNT16, CPED1 |
rs2908007 |
−3.25, 24.67 |
A |
0.60 |
1.08 (1.06–1.10), 1.2×10−20
|
1.05 (1.04–1.06), 5.6×10−22
|
1.06 (1.05–1.07), 2.3×10−39
|
185,055 |
0 |
|
7q21.3 |
C7orf76, SHFM1 |
rs6465508 |
0, 0 |
G |
0.34 |
1.05 (1.03–1.07), 4.0×10−9
|
1.04 (1.03–1.05), 4.1×10−12
|
1.04 (1.03–1.05), 2.0×10−19
|
185,056 |
35 |
|
7p14.1 |
STARD3NL |
rs6959212 |
−89.01 |
T |
0.34 |
1.04 (1.02–1.06), 6.9×10−6
|
1.02 (1.01–1.04), 1.1×10−5
|
1.03 (1.02–1.04), 8.8×10−10
|
185,057 |
15.6 |
|
7p12.1 |
GRB10, COBL |
rs1548607 |
40.33, −182.4 |
G |
0.32 |
1.05 (1.03–1.07), 3.2×10−8
|
1.02 (1.01–1.04), 2.1×10−4
|
1.03 (1.02–1.05), 4.7×10−10
|
185,052 |
40 |
|
9q34.11 |
FUBP3 |
rs7851693 |
0 |
G |
0.35 |
1.03 (1.01–1.06), 1.3×10−4
|
1.05 (1.06–1.06), 4.8×10−16
|
1.04 (1.03–1.05), 5.0×10−19
|
185,057 |
23.5 |
|
10q21.1 |
MBL2/DKK1 |
rs11003047 |
−90.63 |
G |
0.11 |
1.09 (1.07–1.12), 6.2×10−12
|
1.08 (1.07–1.10), 1.4×10−21
|
1.09 (1.07–1.10), 9.5×10−33
|
185,057 |
0 |
|
11q13.2 |
LRP5 |
rs3736228 |
0 |
T |
0.15 |
1.05 (1.03–1.07), 3.0×10−5
|
1.07 (1.05–1.08), 2.8×10−18
|
1.06 (1.05–1.08), 1.0×10−21
|
185,056 |
24.6 |
|
14q32.12 |
RPS6KA5 |
rs1286083 |
0 |
T |
0.82 |
1.04 (1.02–1.06), 8.8×10−5
|
1.05 (1.04–1.07), 3.0×10−14
|
1.05 (1.04–1.06), 1.6×10−17
|
185,085 |
43.3 |
|
17q21.31 |
SOST, DUSP3, MEOX1 |
rs2741856 |
−4.26, −16.65, 88.02 |
G |
0.92 |
1.11 (1.08–1.14), 2.4×10−12 |
1.08 (1.06–1.11), 5.3×10−15
|
1.10 (1.07–1.11), 3.1×10−25
|
184,977 |
0 |
|
18p11.21 |
FAM210A, RNMT |
rs4635400 |
0, −7.149 |
A |
0.36 |
1.06 (1.04–1.07), 1.5×10−12
|
1.03 (1.02–1.04), 2.7×10−9
|
1.04 (1.03–1.05), 1.1×10−18
|
185,057 |
22 |
|
21q22.2 |
ETS2 |
rs9980072 |
141.9 |
G |
0.73 |
1.06 (1.04–1.08), 8.4×10−12
|
1.03 (1.01–1.04), 1.8×10−5
|
1.04 (1.03–1.05), 3.4×10−13
|
185,057 |
36 |
References
- 1. Smith GD, Ebrahim S. Data dredging, bias, or confounding. BMJ 2002;325:1437-1438. https://doi.org/10.1136/bmj.325.7378.1437
- 2. Lawlor DA, Davey Smith G, Kundu D, Bruckdorfer KR, Ebrahim S. Those confounded vitamins: what can we learn from the differences between observational versus randomised trial evidence? Lancet 2004;363:1724-1727. https://doi.org/10.1016/S0140-6736(04)16260-0
- 3. Taubes G. Epidemiology faces its limits. Science 1995;269:164-169. https://doi.org/10.1126/science.7618077
- 4. Sattar N, Preiss D. Reverse causality in cardiovascular epidemiological research: more common than imagined? Circulation 2017;135:2369-2372. https://doi.org/10.1161/CIRCULATIONAHA.117.028307
- 5. Ortola R, Garcia-Esquinas E, Soler-Vila H, Ordovas JM, Lopez-Garcia E, Rodriguez-Artalejo F. Changes in health status predict changes in alcohol consumption in older adults: the Seniors-ENRICA cohort. J Epidemiol Community Health 2019;73:123-129. https://doi.org/10.1136/jech-2018-211104
- 6. Dekkers OM, Vandenbroucke JP, Cevallos M, Renehan AG, Altman DG, Egger M. COSMOS-E: guidance on conducting systematic reviews and meta-analyses of observational studies of etiology. PLoS Med 2019;16:e1002742. https://doi.org/10.1371/journal.pmed.1002742
- 7. Greenland S. An introduction to instrumental variables for epidemiologists. Int J Epidemiol 2000;29:722-729. https://doi.org/10.1093/ije/29.4.722
- 8. Davey Smith G. Capitalizing on Mendelian randomization to assess the effects of treatments. J R Soc Med 2007;100:432-435. https://doi.org/10.1177/014107680710000923
- 9. Davies NM, Howe LJ, Brumpton B, Havdahl A, Evans DM, Davey Smith G. Within family Mendelian randomization studies. Hum Mol Genet 2019;28:R170-R179. https://doi.org/10.1093/hmg/ddz204
- 10. Holmes MV, Dale CE, Zuccolo L, Silverwood RJ, Guo Y, Ye Z, Prieto-Merino D, Dehghan A, Trompet S, Wong A, Cavadino A, Drogan D, Padmanabhan S, Li S, Yesupriya A, Leusink M, Sundstrom J, Hubacek JA, Pikhart H, Swerdlow DI, Panayiotou AG, Borinskaya SA, Finan C, Shah S, Kuchenbaecker KB, Shah T, Engmann J, Folkersen L, Eriksson P, Ricceri F, Melander O, Sacerdote C, Gamble DM, Rayaprolu S, Ross OA, McLachlan S, Vikhireva O, Sluijs I, Scott RA, Adamkova V, Flicker L, Bockxmeer FM, Power C, Marques-Vidal P, Meade T, Marmot MG, Ferro JM, Paulos-Pinheiro S, Humphries SE, Talmud PJ, Mateo Leach I, Verweij N, Linneberg A, Skaaby T, Doevendans PA, Cramer MJ, van der Harst P, Klungel OH, Dowling NF, Dominiczak AF, Kumari M, Nicolaides AN, Weikert C, Boeing H, Ebrahim S, Gaunt TR, Price JF, Lannfelt L, Peasey A, Kubinova R, Pajak A, Malyutina S, Voevoda MI, Tamosiunas A, Maitland-van der Zee AH, Norman PE, Hankey GJ, Bergmann MM, Hofman A, Franco OH, Cooper J, Palmen J, Spiering W, de Jong PA, Kuh D, Hardy R, Uitterlinden AG, Ikram MA, Ford I, Hyppönen E, Almeida OP, Wareham NJ, Khaw KT, Hamsten A, Husemoen LL, Tjønneland A, Tolstrup JS, Rimm E, Beulens JW, Verschuren WM, Onland-Moret NC, Hofker MH, Wannamethee SG, Whincup PH, Morris R, Vicente AM, Watkins H, Farrall M, Jukema JW, Meschia J, Cupples LA, Sharp SJ, Fornage M, Kooperberg C, LaCroix AZ, Dai JY, Lanktree MB, Siscovick DS, Jorgenson E, Spring B, Coresh J, Li YR, Buxbaum SG, Schreiner PJ, Ellison RC, Tsai MY, Patel SR, Redline S, Johnson AD, Hoogeveen RC, Hakonarson H, Rotter JI, Boerwinkle E, de Bakker PI, Kivimaki M, Asselbergs FW, Sattar N, Lawlor DA, Whittaker J, Davey Smith G, Mukamal K, Psaty BM, Wilson JG, Lange LA, Hamidovic A, Hingorani AD, Nordestgaard BG, Bobak M, Leon DA, Langenberg C, Palmer TM, Reiner AP, Keating BJ, Dudbridge F, Casas JP. Association between alcohol and cardiovascular disease: Mendelian randomisation analysis based on individual participant data. BMJ 2014;349:g4164. https://doi.org/10.1136/bmj.g4164
- 11. Gray R, Wheatley K. How to avoid bias when comparing bone marrow transplantation with chemotherapy. Bone Marrow Transplant 1991;7 Suppl 3:9-12.
- 12. Davey Smith G. Capitalising on Mendelian randomization to assess the effects of treatments [Internet]. The James Lind Library; 2006 [cited 2021 Sep 10]. Available from: https://www.jameslindlibrary.org/articles/capitalising-on-mendelian-randomization-to-assess-the-effects-of-treatments/
- 13. Keating S, de Witte T, Suciu S, Willemze R, Hayat M, Labar B, Resegotti L, Ferrini PR, Caronia F, Dardenne M, Solbu G, Petti MC, Vegna ML, Mandelli F, Zittoun RA. The influence of HLA-matched sibling donor availability on treatment outcome for patients with AML: an analysis of the AML 8A study of the EORTC Leukaemia Cooperative Group and GIMEMA. European Organization for Research and Treatment of Cancer. Gruppo Italiano Malattie Ematologiche Maligne dell'Adulto. Br J Haematol 1998;102:1344-1353. https://doi.org/10.1111/j.1365-2141.1998.896hm3674.x
- 14. Bleakley M, Shaw PJ, Nielsen JM. Allogeneic bone marrow transplantation for childhood relapsed acute lymphoblastic leukemia: comparison of outcome in patients with and without a matched family donor. Bone Marrow Transplant 2002;30:1-7. https://doi.org/10.1038/sj.bmt.1703601
- 15. Balduzzi A, Valsecchi MG, Uderzo C, De Lorenzo P, Klingebiel T, Peters C, Stary J, Felice MS, Magyarosy E, Conter V, Reiter A, Messina C, Gadner H, Schrappe M. Chemotherapy versus allogeneic transplantation for very-high-risk childhood acute lymphoblastic leukaemia in first complete remission: comparison by genetic randomisation in an international prospective study. Lancet 2005;366:635-642. https://doi.org/10.1016/S0140-6736(05)66998-X
- 16. Smith GD, Ebrahim S. ‘Mendelian randomization’: can genetic epidemiology contribute to understanding environmental determinants of disease? Int J Epidemiol 2003;32:1-22. https://doi.org/10.1093/ije/dyg070
- 17. Smith GD, Ebrahim S. Mendelian randomization: genetic variants as instruments for strengthening causal inference in observational studies. In: National Research Council (US) Committee on Advances in Collecting and Utilizing Biological Indicators and Genetic Information in Social Science Surveys; Weinstein M, Vaupel JW, Wachter KW, editors. Biosocial surveys [Internet]. National Academies Press (US); 2008 [cited 2021 Sep 10]. Available from: https://www.ncbi.nlm.nih.gov/books/NBK62433/
- 18. Smith GD. Mendelian Randomization for strengthening causal inference in observational studies: application to gene × environment interactions. Perspect Psychol Sci 2010;5:527-545. https://doi.org/10.1177/1745691610383505
- 19. Attermann J, Obel C, Bilenberg N, Nordenbæk CM, Skytthe A, Olsen J. Traits of ADHD and autism in girls with a twin brother: a Mendelian randomization study. Eur Child Adolesc Psychiatry 2012;21:503-509. https://doi.org/10.1007/s00787-012-0287-4
- 20. Sterne JA, Davey Smith G. Sifting the evidence: what’s wrong with significance tests? BMJ 2001;322:226-231. https://doi.org/10.1136/bmj.322.7280.226
- 21. Lawlor DA, Wade KH, Borges MC, Palmer T, Hartwig P, Hemani G, Bowden J. A Mendelian randomization dictionary: useful definitions and descriptions for undertaking, understanding and interpreting Mendelian randomization studies. OSF [Preprint] 2019;Feb 5 https://doi.org/10.31219/osf.io/6yzs7
- 22. Boef AG, Dekkers OM, le Cessie S. Mendelian randomization studies: a review of the approaches used and the quality of reporting. Int J Epidemiol 2015;44:496-511. https://doi.org/10.1093/ije/dyv071
- 23. Lor GC, Risch HA, Fung WT, Au Yeung SL, Wong IO, Zheng W, Pang H. Reporting and guidelines for mendelian randomization analysis: a systematic review of oncological studies. Cancer Epidemiol 2019;62:101577. https://doi.org/10.1016/j.canep.2019.101577
- 24. Diemer EW, Labrecque JA, Neumann A, Tiemeier H, Swanson SA. Mendelian randomisation approaches to the study of prenatal exposures: a systematic review. Paediatr Perinat Epidemiol 2021;35:130-142. https://doi.org/10.1111/ppe.12691
- 25. von Elm E, Altman DG, Egger M, Pocock SJ, Gotzsche PC, Vandenbroucke JP. Strengthening the Reporting of Observational Studies in Epidemiology (STROBE) statement: guidelines for reporting observational studies. BMJ 2007;335:806-808. https://doi.org/10.1136/bmj.39335.541782.AD
- 26. Vandenbroucke JP, von Elm E, Altman DG, Gotzsche PC, Mulrow CD, Pocock SJ, Poole C, Schlesselman JJ, Egger M. Strengthening the Reporting of Observational Studies in Epidemiology (STROBE): explanation and elaboration. PLoS Med 2007;4:e297. https://doi.org/10.1371/journal.pmed.0040297
- 27. Skrivankova VW, Richmond RC, Woolf BA, Yarmolinsky J, Davies NM, Swanson SA, VanderWeele TJ, Higgins JP, Timpson NJ, Dimou N, Langenberg C, Golub RM, Loder EW, Gallo V, Tybjaerg-Hansen A, Davey Smith G, Egger M, Richards JB. Strengthening the Reporting of Observational Studies in Epidemiology using Mendelian randomization: the STROBE-MR statement. JAMA 2021;326:1614-1621. https://doi.org/10.1001/jama.2021.18236
- 28. Moher D, Schulz KF, Simera I, Altman DG. Guidance for developers of health research reporting guidelines. PLoS Med 2010;7:e1000217. https://doi.org/10.1371/journal.pmed.1000217
- 29. Smith GD, Davies NM, Dimou N, Egger M, Gallo V, Golub R, Higgins JP, Langenberg C, Loder EW, Richards JB, Richmond RC. STROBE-MR: guidelines for strengthening the reporting of Mendelian randomization studies. PeerJ [Preprint] 2019;Jul 15 https://doi.org/10.7287/peerj.preprints.27857v1
- 30. University of Bristol; MRC Integrative Epidemiology Unit. 4th Mendelian Randomization Conference 2019 [Internet]. University of Bristol; 2020 [cited 2021 Sep 10]. Available from: https://www.bristol.ac.uk/integrative-epidemiology/seminars/2019/mr-conference-2019.html
- 31. Burgess S, Davey Smith G, Davies NM, Dudbridge F, Gill D, Glymour MM, Hartwig FP, Kutalik Z, Holmes MV, Minelli C, Morrison JV, Pan W, Relton CL, Theodoratou E. Guidelines for performing Mendelian randomization investigations: update for summer 2023. Wellcome Open Res 2019;4:186. https://doi.org/10.12688/wellcomeopenres.15555.3
- 32. Corbin LJ, Richmond RC, Wade KH, Burgess S, Bowden J, Smith GD, Timpson NJ. BMI as a modifiable risk factor for type 2 diabetes: refining and understanding causal estimates using Mendelian randomization. Diabetes 2016;65:3002-3007. https://doi.org/10.2337/db16-0418
- 33. Ligthart S, Vaez A, Vosa U, Stathopoulou MG, de Vries PS, Prins BP, Van der Most PJ, Tanaka T, Naderi E, Rose LM, Wu Y, Karlsson R, Barbalic M, Lin H, Pool R, Zhu G, Mace A, Sidore C, Trompet S, Mangino M, Sabater-Lleal M, Kemp JP, Abbasi A, Kacprowski T, Verweij N, Smith AV, Huang T, Marzi C, Feitosa MF, Lohman KK, Kleber ME, Milaneschi Y, Mueller C, Huq M, Vlachopoulou E, Lyytikainen LP, Oldmeadow C, Deelen J, Perola M, Zhao JH, Feenstra B, Amini M, Lahti J, Schraut KE, Fornage M, Suktitipat B, Chen WM, Li X, Nutile T, Malerba G, Luan J, Bak T, Schork N, Del Greco M F, Thiering E, Mahajan A, Marioni RE, Mihailov E, Eriksson J, Ozel AB, Zhang W, Nethander M, Cheng YC, Aslibekyan S, Ang W, Gandin I, Yengo L, Portas L, Kooperberg C, Hofer E, Rajan KB, Schurmann C, den Hollander W, Ahluwalia TS, Zhao J, Draisma HH, Ford I, Timpson N, Teumer A, Huang H, Wahl S, Liu Y, Huang J, Uh HW, Geller F, Joshi PK, Yanek LR, Trabetti E, Lehne B, Vozzi D, Verbanck M, Biino G, Saba Y, Meulenbelt I, O'Connell JR, Laakso M, Giulianini F, Magnusson PK, Ballantyne CM, Hottenga JJ, Montgomery GW, Rivadineira F, Rueedi R, Steri M, Herzig KH, Stott DJ, Menni C, Franberg M, St Pourcain B, Felix SB, Pers TH, Bakker SJ, Kraft P, Peters A, Vaidya D, Delgado G, Smit JH, Großmann V, Sinisalo J, Seppala I, Williams SR, Holliday EG, Moed M, Langenberg C, Raikkonen K, Ding J, Campbell H, Sale MM, Chen YI, James AL, Ruggiero D, Soranzo N, Hartman CA, Smith EN, Berenson GS, Fuchsberger C, Hernandez D, Tiesler CM, Giedraitis V, Liewald D, Fischer K, Mellstrom D, Larsson A, Wang Y, Scott WR, Lorentzon M, Beilby J, Ryan KA, Pennell CE, Vuckovic D, Balkau B, Concas MP, Schmidt R, Mendes de Leon CF, Bottinger EP, Kloppenburg M, Paternoster L, Boehnke M, Musk AW, Willemsen G, Evans DM, Madden PA, Kahonen M, Kutalik Z, Zoledziewska M, Karhunen V, Kritchevsky SB, Sattar N, Lachance G, Clarke R, Harris TB, Raitakari OT, Attia JR, van Heemst D, Kajantie E, Sorice R, Gambaro G, Scott RA, Hicks AA, Ferrucci L, Standl M, Lindgren CM, Starr JM, Karlsson M, Lind L, Li JZ, Chambers JC, Mori TA, de Geus EJ, Heath AC, Martin NG, Auvinen J, Buckley BM, de Craen AJM, Waldenberger M, Strauch K, Meitinger T, Scott RJ, McEvoy M, Beekman M, Bombieri C, Ridker PM, Mohlke KL, Pedersen NL, Morrison AC, Boomsma DI, Whitfield JB, Strachan DP, Hofman A, Vollenweider P, Cucca F, Jarvelin MR, Jukema JW, Spector TD, Hamsten A, Zeller T, Uitterlinden AG, Nauck M, Gudnason V, Qi L, Grallert H, Borecki IB, Rotter JI, Marz W, Wild PS, Lokki ML, Boyle M, Salomaa V, Melbye M, Eriksson JG, Wilson JF, Penninx BW, Becker DM, Worrall BB, Gibson G, Krauss RM, Ciullo M, Zaza G, Wareham NJ, Oldehinkel AJ, Palmer LJ, Murray SS, Pramstaller PP, Bandinelli S, Heinrich J, Ingelsson E, Deary IJ, Mägi R, Vandenput L, van der Harst P, Desch KC, Kooner JS, Ohlsson C, Hayward C, Lehtimäki T, Shuldiner AR, Arnett DK, Beilin LJ, Robino A, Froguel P, Pirastu M, Jess T, Koenig W, Loos RJ, Evans DA, Schmidt H, Smith GD, Slagboom PE, Eiriksdottir G, Morris AP, Psaty BM, Tracy RP, Nolte IM, Boerwinkle E, Visvikis-Siest S, Reiner AP, Gross M, Bis JC, Franke L, Franco OH, Benjamin EJ, Chasman DI, Dupuis J, Snieder H, Dehghan A, Alizadeh BZ. Genome analyses of >200,000 individuals identify 58 loci for chronic inflammation and highlight pathways that link inflammation and complex disorders. Am J Hum Genet 2018;103:691-706. https://doi.org/10.1016/j.ajhg.2018.09.009
- 34. Burgess S, Ference BA, Staley JR, Freitag DF, Mason AM, Nielsen SF, Willeit P, Young R, Surendran P, Karthikeyan S, Bolton TR, Peters JE, Kamstrup PR, Tybjærg-Hansen A, Benn M, Langsted A, Schnohr P, Vedel-Krogh S, Kobylecki CJ, Ford I, Packard C, Trompet S, Jukema JW, Sattar N, Di Angelantonio E, Saleheen D, Howson JMM, Nordestgaard BG, Butterworth AS, Danesh J. Association of LPA variants with risk of coronary disease and the implications for lipoprotein(a)-lowering therapies: a Mendelian randomization analysis. JAMA Cardiol 2018;3:619-627. https://doi.org/10.1001/jamacardio.2018.1470
- 35. Harroud A, Morris JA, Forgetta V, Mitchell R, Smith GD, Sawcer SJ, Richards JB. Effect of age at puberty on risk of multiple sclerosis: a Mendelian randomization study. Neurology 2019;92:e1803-e1810. https://doi.org/10.1212/WNL.0000000000007325
- 36. Burgess S, Davies NM, Thompson SG. Instrumental variable analysis with a nonlinear exposure-outcome relationship. Epidemiology 2014;25:877-885. https://doi.org/10.1097/EDE.0000000000000161
- 37. Zheng J, Haberland V, Baird D, Walker V, Haycock PC, Hurle MR, Gutteridge A, Erola P, Liu Y, Luo S, Robinson J, Richardson TG, Staley JR, Elsworth B, Burgess S, Sun BB, Danesh J, Runz H, Maranville JC, Martin HM, Yarmolinsky J, Laurin C, Holmes MV, Liu JZ, Estrada K, Santos R, McCarthy L, Waterworth D, Nelson MR, Smith GD, Butterworth AS, Hemani G, Scott RA, Gaunt TR. Phenome-wide Mendelian randomization mapping the influence of the plasma proteome on complex diseases. Nat Genet 2020;52:1122-1131. https://doi.org/10.1038/s41588-020-0682-6
- 38. Larsson SC, Burgess S, Michaelsson K. Association of genetic variants related to serum calcium levels with coronary artery disease and myocardial infarction. JAMA 2017;318:371-380. https://doi.org/10.1001/jama.2017.8981
- 39. Wootton RE, Lawn RB, Millard LAC, Davies NM, Taylor AE, Munafo MR, Timpson NJ, Davis OS, Davey Smith G, Haworth CM. Evaluation of the causal effects between subjective wellbeing and cardiometabolic health: Mendelian randomisation study. BMJ 2018;362:k3788. https://doi.org/10.1136/bmj.k3788
- 40. Vimaleswaran KS, Berry DJ, Lu C, Tikkanen E, Pilz S, Hiraki LT, Cooper JD, Dastani Z, Li R, Houston DK, Wood AR, Michaelsson K, Vandenput L, Zgaga L, Yerges-Armstrong LM, McCarthy MI, Dupuis J, Kaakinen M, Kleber ME, Jameson K, Arden N, Raitakari O, Viikari J, Lohman KK, Ferrucci L, Melhus H, Ingelsson E, Byberg L, Lind L, Lorentzon M, Salomaa V, Campbell H, Dunlop M, Mitchell BD, Herzig KH, Pouta A, Hartikainen AL, Streeten EA, Theodoratou E, Jula A, Wareham NJ, Ohlsson C, Frayling TM, Kritchevsky SB, Spector TD, Richards JB, Lehtimaki T, Ouwehand WH, Kraft P, Cooper C, Marz W, Power C, Loos RJ, Wang TJ, Jarvelin MR, Whittaker JC, Hingorani AD, Hypponen E. Causal relationship between obesity and vitamin D status: bi-directional Mendelian randomization analysis of multiple cohorts. PLoS Med 2013;10:e1001383. https://doi.org/10.1371/journal.pmed.1001383
- 41. Trajanoska K, Morris JA, Oei L, Zheng HF, Evans DM, Kiel DP, Ohlsson C, Richards JB, Rivadeneira F. Assessment of the genetic and clinical determinants of fracture risk: genome wide association and mendelian randomisation study. BMJ 2018;362:k3225. https://doi.org/10.1136/bmj.k3225
- 42. Tyrrell J, Jones SE, Beaumont R, Astley CM, Lovell R, Yaghootkar H, Tuke M, Ruth KS, Freathy RM, Hirschhorn JN, Wood AR, Murray A, Weedon MN, Frayling TM. Height, body mass index, and socioeconomic status: mendelian randomisation study in UK Biobank. BMJ 2016;352:i582. https://doi.org/10.1136/bmj.i582
- 43. Brion MJ, Shakhbazov K, Visscher PM. Calculating statistical power in Mendelian randomization studies. Int J Epidemiol 2013;42:1497-1501. https://doi.org/10.1093/ije/dyt179
- 44. Brion MJ, Shakhbazov K, Visscher PM. mRnd: power calculations for Mendelian randomization [Internet]. University of Queensland, Institute for Molecular Bioscience; 2013 [cited 2021 Sep 10] Available from: https://shiny.cnsgenomics.com/mRnd/
- 45. Morris JA, Kemp JP, Youlten SE, Laurent L, Logan JG, Chai RC, Vulpescu NA, Forgetta V, Kleinman A, Mohanty ST, Sergio CM, Quinn J, Nguyen-Yamamoto L, Luco AL, Vijay J, Simon MM, Pramatarova A, Medina-Gomez C, Trajanoska K, Ghirardello EJ, Butterfield NC, Curry KF, Leitch VD, Sparkes PC, Adoum AT, Mannan NS, Komla-Ebri DS, Pollard AS, Dewhurst HF, Hassall TA, Beltejar MG, Adams DJ, Vaillancourt SM, Kaptoge S, Baldock P, Cooper C, Reeve J, Ntzani EE, Evangelou E, Ohlsson C, Karasik D, Rivadeneira F, Kiel DP, Tobias JH, Gregson CL, Harvey NC, Grundberg E, Goltzman D, Adams DJ, Lelliott CJ, Hinds DA, Ackert-Bicknell CL, Hsu YH, Maurano MT, Croucher PI, Williams GR, Bassett JH, Evans DM, Richards JB. An atlas of genetic influences on osteoporosis in humans and mice. Nat Genet 2019;51:258-266. https://doi.org/10.1038/s41588-018-0302-x
- 46. Johansson M, Carreras-Torres R, Scelo G, Purdue MP, Mariosa D, Muller DC, Timpson NJ, Haycock PC, Brown KM, Wang Z, Ye Y, Hofmann JN, Foll M, Gaborieau V, Machiela MJ, Colli LM, Li P, Garnier JG, Blanche H, Boland A, Burdette L, Prokhortchouk E, Skryabin KG, Yeager M, Radojevic-Skodric S, Ognjanovic S, Foretova L, Holcatova I, Janout V, Mates D, Mukeriya A, Rascu S, Zaridze D, Bencko V, Cybulski C, Fabianova E, Jinga V, Lissowska J, Lubinski J, Navratilova M, Rudnai P, Benhamou S, Cancel-Tassin G, Cussenot O, Weiderpass E, Ljungberg B, Tumkur Sitaram R, Haggstrom C, Bruinsma F, Jordan SJ, Severi G, Winship I, Hveem K, Vatten LJ, Fletcher T, Larsson SC, Wolk A, Banks RE, Selby PJ, Easton DF, Andreotti G, Beane Freeman LE, Koutros S, Männistö S, Weinstein S, Clark PE, Edwards TL, Lipworth L, Gapstur SM, Stevens VL, Carol H, Freedman ML, Pomerantz MM, Cho E, Wilson KM, Gaziano JM, Sesso HD, Freedman ND, Parker AS, Eckel-Passow JE, Huang WY, Kahnoski RJ, Lane BR, Noyes SL, Petillo D, Teh BT, Peters U, White E, Anderson GL, Johnson L, Luo J, Buring J, Lee IM, Chow WH, Moore LE, Eisen T, Henrion M, Larkin J, Barman P, Leibovich BC, Choueiri TK, Lathrop GM, Deleuze JF, Gunter M, McKay JD, Wu X, Houlston RS, Chanock SJ, Relton C, Richards JB, Martin RM, Davey Smith G, Brennan P. The influence of obesity-related factors in the etiology of renal cell carcinoma: a Mendelian randomization study. PLoS Med 2019;16:e1002724. https://doi.org/10.1371/journal.pmed.1002724
- 47. Burgess S, Scott RA, Timpson NJ, Davey Smith G, Thompson SG. Using published data in Mendelian randomization: a blueprint for efficient identification of causal risk factors. Eur J Epidemiol 2015;30:543-552. https://doi.org/10.1007/s10654-015-0011-z
- 48. Lotta LA, Wittemans LB, Zuber V, Stewart ID, Sharp SJ, Luan J, Day FR, Li C, Bowker N, Cai L, De Lucia Rolfe E, Khaw KT, Perry JRB, O'Rahilly S, Scott RA, Savage DB, Burgess S, Wareham NJ, Langenberg C. Association of genetic variants related to gluteofemoral vs abdominal fat distribution with type 2 diabetes, coronary disease, and cardiovascular risk factors. JAMA 2018;320:2553-2563. https://doi.org/10.1001/jama.2018.19329
- 49. Tyrrell J, Richmond RC, Palmer TM, Feenstra B, Rangarajan J, Metrustry S, Cavadino A, Paternoster L, Armstrong LL, De Silva NM, Wood AR, Horikoshi M, Geller F, Myhre R, Bradfield JP, Kreiner-Moller E, Huikari V, Painter JN, Hottenga JJ, Allard C, Berry DJ, Bouchard L, Das S, Evans DM, Hakonarson H, Hayes MG, Heikkinen J, Hofman A, Knight B, Lind PA, McCarthy MI, McMahon G, Medland SE, Melbye M, Morris AP, Nodzenski M, Reichetzeder C, Ring SM, Sebert S, Sengpiel V, Sorensen TI, Willemsen G, de Geus EJ, Martin NG, Spector TD, Power C, Jarvelin MR, Bisgaard H, Grant SF, Nohr EA, Jaddoe VW, Jacobsson B, Murray JC, Hocher B, Hattersley AT, Scholtens DM, Davey Smith G, Hivert MF, Felix JF, Hypponen E, Lowe WL, Frayling TM, Lawlor DA, Freathy RM. Genetic evidence for causal relationships between maternal obesity-related traits and birth weight. JAMA 2016;315:1129-1140. https://doi.org/10.1001/jama.2016.1975
- 50. World Medical Association. World Medical Association Declaration of Helsinki: ethical principles for medical research involving human subjects. JAMA 2013;310:2191-2194. https://doi.org/10.1001/jama.2013.281053
- 51. Carreras-Torres R, Johansson M, Haycock PC, Relton CL, Davey Smith G, Brennan P, Martin RM. Role of obesity in smoking behaviour: Mendelian randomisation study in UK Biobank. BMJ 2018;361:k1767. https://doi.org/10.1136/bmj.k1767
- 52. Mokry LE, Ross S, Timpson NJ, Sawcer S, Davey Smith G, Richards JB. Obesity and multiple sclerosis: a Mendelian randomization study. PLoS Med 2016;13:e1002053. https://doi.org/10.1371/journal.pmed.1002053
- 53. Swanson SA, Hernan MA, Miller M, Robins JM, Richardson TS. Partial identification of the average treatment effect using instrumental variables: review of methods for binary instruments, treatments, and outcomes. J Am Stat Assoc 2018;113:933-947. https://doi.org/10.1080/01621459.2018.1434530
- 54. Angrist JD, Imbens GW, Rubin DB. Identification of causal effects using instrumental variables. J Am Stat Assoc 1996;91:444-455. https://doi.org/10.1080/01621459.1996.10476902
- 55. Bowden J, Davey Smith G, Burgess S. Mendelian randomization with invalid instruments: effect estimation and bias detection through Egger regression. Int J Epidemiol 2015;44:512-525. https://doi.org/10.1093/ije/dyv080
- 56. Bowden J, Davey Smith G, Haycock PC, Burgess S. Consistent estimation in Mendelian randomization with some invalid instruments using a weighted median estimator. Genet Epidemiol 2016;40:304-314. https://doi.org/10.1002/gepi.21965
- 57. Hartwig FP, Davey Smith G, Bowden J. Robust inference in summary data Mendelian randomization via the zero modal pleiotropy assumption. Int J Epidemiol 2017;46:1985-1998. https://doi.org/10.1093/ije/dyx102
- 58. Davies NM, Holmes MV, Davey Smith G. Reading Mendelian randomisation studies: a guide, glossary, and checklist for clinicians. BMJ 2018;362:k601. https://doi.org/10.1136/bmj.k601
- 59. VanderWeele TJ, Tchetgen Tchetgen EJ, Cornelis M, Kraft P. Methodological challenges in Mendelian randomization. Epidemiology 2014;25:427-435. https://doi.org/10.1097/EDE.0000000000000081
- 60. Swanson SA, Tiemeier H, Ikram MA, Hernan MA. Nature as a trialist?: deconstructing the analogy between Mendelian randomization and randomized trials. Epidemiology 2017;28:653-659. https://doi.org/10.1097/EDE.0000000000000699
- 61. Swanson SA, Hernan MA. The challenging interpretation of instrumental variable estimates under monotonicity. Int J Epidemiol 2018;47:1289-1297. https://doi.org/10.1093/ije/dyx038
- 62. Hartwig FP, Davies NM, Davey Smith G. Bias in Mendelian randomization due to assortative mating. Genet Epidemiol 2018;42:608-620. https://doi.org/10.1002/gepi.22138
- 63. Brumpton B, Sanderson E, Heilbron K, Hartwig FP, Harrison S, Vie GA, Cho Y, Howe LD, Hughes A, Boomsma DI, Havdahl A, Hopper J, Neale M, Nivard MG, Pedersen NL, Reynolds CA, Tucker-Drob EM, Grotzinger A, Howe L, Morris T, Li S, Auton A, Windmeijer F, Chen WM, Bjørngaard JH, Hveem K, Willer C, Evans DM, Kaprio J, Davey Smith G, Asvold BO, Hemani G, Davies NM. Avoiding dynastic, assortative mating, and population stratification biases in Mendelian randomization through within-family analyses. Nat Commun 2020;11:3519. https://doi.org/10.1038/s41467-020-17117-4
- 64. Swanson SA. A practical guide to selection bias in instrumental variable analyses. Epidemiology 2019;30:345-349. https://doi.org/10.1097/EDE.0000000000000973
- 65. Bound J, Jaeger DA, Baker RM. Problems with instrumental variables estimation when the correlation between the instruments and the endogenous explanatory variable is weak. J Am Stat Assoc 1995;90:443-450. https://doi.org/10.1080/01621459.1995.10476536
- 66. Sanderson E, Richardson TG, Hemani G, Davey Smith G. The use of negative control outcomes in Mendelian randomization to detect potential population stratification. Int J Epidemiol 2021;50:1350-1361. https://doi.org/10.1093/ije/dyaa288
- 67. Brookhart MA, Rassen JA, Schneeweiss S. Instrumental variable methods in comparative safety and effectiveness research. Pharmacoepidemiol Drug Saf 2010;19:537-554. https://doi.org/10.1002/pds.1908
- 68. Jackson JW, Swanson SA. Toward a clearer portrayal of confounding bias in instrumental variable applications. Epidemiology 2015;26:498-504. https://doi.org/10.1097/EDE.0000000000000287
- 69. Swanson SA, VanderWeele TJ. E-values for Mendelian randomization. Epidemiology 2020;31:e23-e24. https://doi.org/10.1097/EDE.0000000000001164
- 70. Davies NM, Thomas KH, Taylor AE, Taylor GMJ, Martin RM, Munafo MR, Windmeijer F. How to compare instrumental variable and conventional regression analyses using negative controls and bias plots. Int J Epidemiol 2017;46:2067-2077. https://doi.org/10.1093/ije/dyx014
- 71. Hernan MA, Robins JM. Estimating causal effects from epidemiological data. J Epidemiol Community Health 2006;60:578-586. https://doi.org/10.1136/jech.2004.029496
- 72. Mills HL, Higgins JPT, Morris RW, Kessler D, Heron J, Wiles N, Davey Smith G, Tilling K. Detecting heterogeneity of intervention effects using analysis and meta-analysis of differences in variance between trial arms. Epidemiology 2021;32:846-854. https://doi.org/10.1097/EDE.0000000000001401
- 73. Glymour MM, Tchetgen Tchetgen EJ, Robins JM. Credible Mendelian randomization studies: approaches for evaluating the instrumental variable assumptions. Am J Epidemiol 2012;175:332-339. https://doi.org/10.1093/aje/kwr323
- 74. Burgess S, Bowden J, Fall T, Ingelsson E, Thompson SG. Sensitivity analyses for robust causal inference from Mendelian randomization analyses with multiple genetic variants. Epidemiology 2017;28:30-42. https://doi.org/10.1097/EDE.0000000000000559
- 75. VanderWeele TJ, Ding P. Sensitivity analysis in observational research: introducing the E-value. Ann Intern Med 2017;167:268-274. https://doi.org/10.7326/M16-2607
- 76. Conley TG, Hansen CB, Rossi PE. Plausibly exogenous. Rev Econ Stat 2012;94:260-272. https://doi.org/10.1162/REST_a_00139
- 77. Labrecque JA, Swanson SA. Interpretation and potential biases of mendelian randomization estimates with time-varying exposures. Am J Epidemiol 2019;188:231-238. https://doi.org/10.1093/aje/kwy204
- 78. Mokry LE, Ross S, Ahmad OS, Forgetta V, Smith GD, Goltzman D, Leong A, Greenwood CM, Thanassoulis G, Richards JB. Vitamin D and risk of multiple sclerosis: a Mendelian randomization study. PLoS Med 2015;12:e1001866. https://doi.org/10.1371/journal.pmed.1001866
- 79. Millard LAC, Davies NM, Tilling K, Gaunt TR, Davey Smith G. Searching for the causal effects of body mass index in over 300 000 participants in UK Biobank, using Mendelian randomization. PLoS Genet 2019;15:e1007951. https://doi.org/10.1371/journal.pgen.1007951
- 80. Burgess S, Thompson SG. Use of allele scores as instrumental variables for Mendelian randomization. Int J Epidemiol 2013;42:1134-1144. https://doi.org/10.1093/ije/dyt093
- 81. Zhao JV, Luo S, Schooling CM. Sex-specific Mendelian randomization study of genetically predicted insulin and cardiovascular events in the UK Biobank. Commun Biol 2019;2:332. https://doi.org/10.1038/s42003-019-0579-z
- 82. Burgess S, Seaman S, Lawlor DA, Casas JP, Thompson SG. Missing data methods in Mendelian randomization studies with multiple instruments. Am J Epidemiol 2011;174:1069-1076. https://doi.org/10.1093/aje/kwr235
- 83. Liyanage UE, Ong JS, An J, Gharahkhani P, Law MH, MacGregor S. Mendelian randomization study for genetically predicted polyunsaturated fatty acids levels on overall cancer risk and mortality. Cancer Epidemiol Biomarkers Prev 2019;28:1015-1023. https://doi.org/10.1158/1055-9965.EPI-18-0940
- 84. Hemani G, Zheng J, Elsworth B, Wade KH, Haberland V, Baird D, Laurin C, Burgess S, Bowden J, Langdon R, Tan VY, Yarmolinsky J, Shihab HA, Timpson NJ, Evans DM, Relton C, Martin RM, Davey Smith G, Gaunt TR, Haycock PC. The MR-Base platform supports systematic causal inference across the human phenome. Elife 2018;7:e34408. https://doi.org/10.7554/eLife.34408
- 85. Schmidt AF, Finan C, Gordillo-Maranon M, Asselbergs FW, Freitag DF, Patel RS, Tyl B, Chopade S, Faraway R, Zwierzyna M, Hingorani AD. Genetic drug target validation using Mendelian randomisation. Nat Commun 2020;11:3255. https://doi.org/10.1038/s41467-020-16969-0
- 86. Labrecque J, Swanson SA. Understanding the assumptions underlying instrumental variable analyses: a brief review of falsification strategies and related tools. Curr Epidemiol Rep 2018;5:214-220. https://doi.org/10.1007/s40471-018-0152-1
- 87. Mountjoy E, Davies NM, Plotnikov D, Smith GD, Rodriguez S, Williams CE, Guggenheim JA, Atan D. Education and myopia: assessing the direction of causality by Mendelian randomisation. BMJ 2018;361:k2022. https://doi.org/10.1136/bmj.k2022
- 88. Pasman JA, Verweij KJ, Gerring Z, Stringer S, Sanchez-Roige S, Treur JL, Abdellaoui A, Nivard MG, Baselmans BM, Ong JS, Ip HF, van der Zee MD, Bartels M, Day FR, Fontanillas P, Elson SL, de Wit H, Davis LK, MacKillop J, Derringer JL, Branje SJ, Hartman CA, Heath AC, van Lier PA, Madden PA, Magi R, Meeus W, Montgomery GW, Oldehinkel AJ, Pausova Z, Ramos-Quiroga JA, Paus T, Ribases M, Kaprio J, Boks MP, Bell JT, Spector TD, Gelernter J, Boomsma DI, Martin NG, MacGregor S, Perry JR, Palmer AA, Posthuma D, Munafo MR, Gillespie NA, Derks EM, Vink JM. GWAS of lifetime cannabis use reveals new risk loci, genetic overlap with psychiatric traits, and a causal influence of schizophrenia. Nat Neurosci 2018;21:1161-1170. https://doi.org/10.1038/s41593-018-0206-1
- 89. Carter AR, Gill D, Davies NM, Taylor AE, Tillmann T, Vaucher J, Wootton RE, Munafo MR, Hemani G, Malik R, Seshadri S, Woo D, Burgess S, Davey Smith G, Holmes MV, Tzoulaki I, Howe LD, Dehghan A. Understanding the consequences of education inequality on cardiovascular disease: Mendelian randomisation study. BMJ 2019;365:l1855. https://doi.org/10.1136/bmj.l1855
- 90. Baldwin JR, Pingault JB, Schoeler T, Sallis HM, Munafo MR. Protecting against researcher bias in secondary data analysis: challenges and potential solutions. Eur J Epidemiol 2022;37:1-10. https://doi.org/10.1007/s10654-021-00839-0
- 91. Higgins JP, Thompson SG. Quantifying heterogeneity in a meta-analysis. Stat Med 2002;21:1539-1558. https://doi.org/10.1002/sim.1186
- 92. Ioannidis JP, Patsopoulos NA, Evangelou E. Uncertainty in heterogeneity estimates in meta-analyses. BMJ 2007;335:914-916. https://doi.org/10.1136/bmj.39343.408449.80
- 93. Mhatre S, Richmond RC, Chatterjee N, Rajaraman P, Wang Z, Zhang H, Badwe R, Goel M, Patkar S, Shrikhande SV, Patil PS, Davey Smith G, Relton CL, Dikshit RP. The role of gallstones in gallbladder cancer in India: a Mendelian randomization study. Cancer Epidemiol Biomarkers Prev 2021;30:396-403. https://doi.org/10.1158/1055-9965.EPI-20-0919
- 94. Burgess S, Davies NM, Thompson SG. Bias due to participant overlap in two-sample Mendelian randomization. Genet Epidemiol 2016;40:597-608. https://doi.org/10.1002/gepi.21998
- 95. Garner C. Upward bias in odds ratio estimates from genome-wide association studies. Genet Epidemiol 2007;31:288-295. https://doi.org/10.1002/gepi.20209
- 96. Gill D, Zuber V, Dawson J, Pearson-Stuttard J, Carter AR, Sanderson E, Karhunen V, Levin MG, Wootton RE, Klarin D, Tsao PS, Tsilidis KK, Damrauer SM, Burgess S, Elliott P. Risk factors mediating the effect of body mass index and waist-to-hip ratio on cardiovascular outcomes: Mendelian randomization analysis. Int J Obes (Lond) 2021;45:1428-1438. https://doi.org/10.1038/s41366-021-00807-4
- 97. Lotta LA, Sharp SJ, Burgess S, Perry JR, Stewart ID, Willems SM, Luan J, Ardanaz E, Arriola L, Balkau B, Boeing H, Deloukas P, Forouhi NG, Franks PW, Grioni S, Kaaks R, Key TJ, Navarro C, Nilsson PM, Overvad K, Palli D, Panico S, Quiros JR, Riboli E, Rolandsson O, Sacerdote C, Salamanca EC, Slimani N, Spijkerman AM, Tjonneland A, Tumino R, van der A DL, van der Schouw YT, McCarthy MI, Barroso I, O'Rahilly S, Savage DB, Sattar N, Langenberg C, Scott RA, Wareham NJ. Association between low-density lipoprotein cholesterol-lowering genetic variants and risk of type 2 diabetes: a meta-analysis. JAMA 2016;316:1383-1391. https://doi.org/10.1001/jama.2016.14568
- 98. Lane JM, Jones SE, Dashti HS, Wood AR, Aragam KG, van Hees VT, Strand LB, Winsvold BS, Wang H, Bowden J, Song Y, Patel K, Anderson SG, Beaumont RN, Bechtold DA, Cade BE, Haas M, Kathiresan S, Little MA, Luik AI, Loudon AS, Purcell S, Richmond RC, Scheer FA, Schormair B, Tyrrell J, Winkelman JW, Winkelmann J, Hveem K, Zhao C, Nielsen JB, Willer CJ, Redline S, Spiegelhalder K, Kyle SD, Ray DW, Zwart JA, Brumpton B, Frayling TM, Lawlor DA, Rutter MK, Weedon MN, Saxena R. Biological and clinical insights from genetics of insomnia symptoms. Nat Genet 2019;51:387-393. https://doi.org/10.1038/s41588-019-0361-7
- 99. Sun YQ, Burgess S, Staley JR, Wood AM, Bell S, Kaptoge SK, Guo Q, Bolton TR, Mason AM, Butterworth AS, Di Angelantonio E, Vie GA, Bjorngaard JH, Kinge JM, Chen Y, Mai XM. Body mass index and all cause mortality in HUNT and UK Biobank studies: linear and non-linear mendelian randomisation analyses. BMJ 2019;364:l1042. https://doi.org/10.1136/bmj.l1042
- 100. Bowden J, Del Greco M F, Minelli C, Davey Smith G, Sheehan N, Thompson J. A framework for the investigation of pleiotropy in two-sample summary data Mendelian randomization. Stat Med 2017;36:1783-1802. https://doi.org/10.1002/sim.7221
- 101. Larsson SC, Back M, Rees JM, Mason AM, Burgess S. Body mass index and body composition in relation to 14 cardiovascular conditions in UK Biobank: a Mendelian randomization study. Eur Heart J 2020;41:221-226. https://doi.org/10.1093/eurheartj/ehz388
- 102. Davey Smith G, Hemani G. Mendelian randomization: genetic anchors for causal inference in epidemiological studies. Hum Mol Genet 2014;23:R89-R98. https://doi.org/10.1093/hmg/ddu328
- 103. Munafo MR, Davey Smith G. Robust research needs many lines of evidence. Nature 2018;553:399-401. https://doi.org/10.1038/d41586-018-01023-3
- 104. Lawlor DA, Tilling K, Davey Smith G. Triangulation in aetiological epidemiology. Int J Epidemiol 2016;45:1866-1886. https://doi.org/10.1093/ije/dyw314
- 105. Bowden J, Spiller W, Del Greco M F, Sheehan N, Thompson J, Minelli C, Davey Smith G. Improving the visualization, interpretation and analysis of two-sample summary data Mendelian randomization via the Radial plot and Radial regression. Int J Epidemiol 2018;47:1264-1278. https://doi.org/10.1093/ije/dyy101
- 106. Hess DR. How to write an effective discussion. Respir Care 2004;49:1238-1241.
- 107. Docherty M, Smith R. The case for structuring the discussion of scientific papers. BMJ 1999;318:1224-1225. https://doi.org/10.1136/bmj.318.7193.1224
- 108. Perneger TV, Hudelson PM. Writing a research article: advice to beginners. Int J Qual Health Care 2004;16:191-192. https://doi.org/10.1093/intqhc/mzh053
- 109. Ference BA, Kastelein JJ, Ginsberg HN, Chapman MJ, Nicholls SJ, Ray KK, Packard CJ, Laufs U, Brook RD, Oliver-Williams C, Butterworth AS, Danesh J, Smith GD, Catapano AL, Sabatine MS. Association of genetic variants related to CETP inhibitors and statins with lipoprotein levels and cardiovascular risk. JAMA 2017;318:947-956. https://doi.org/10.1001/jama.2017.11467
- 110. Cole SR, Frangakis CE. The consistency statement in causal inference: a definition or an assumption? Epidemiology 2009;20:3-5. https://doi.org/10.1097/EDE.0b013e31818ef366
- 111. Richardson TG, Sanderson E, Elsworth B, Tilling K, Davey Smith G. Use of genetic variation to separate the effects of early and later life adiposity on disease risk: Mendelian randomisation study. BMJ 2020;369:m1203. https://doi.org/10.1136/bmj.m1203
- 112. Holmes MV, Smith GD. Dyslipidaemia: revealing the effect of CETP inhibition in cardiovascular disease. Nat Rev Cardiol 2017;14:635-636. https://doi.org/10.1038/nrcardio.2017.156
- 113. Tillmann T, Vaucher J, Okbay A, Pikhart H, Peasey A, Kubinova R, Pajak A, Tamosiunas A, Malyutina S, Hartwig FP, Fischer K, Veronesi G, Palmer T, Bowden J, Davey Smith G, Bobak M, Holmes MV. Education and coronary heart disease: Mendelian randomisation study. BMJ 2017;358:j3542. https://doi.org/10.1136/bmj.j3542
- 114. Burgess S, Labrecque JA. Mendelian randomization with a binary exposure variable: interpretation and presentation of causal estimates. Eur J Epidemiol 2018;33:947-952. https://doi.org/10.1007/s10654-018-0424-6
- 115. Daghlas I, Guo Y, Chasman DI. Effect of genetic liability to migraine on coronary artery disease and atrial fibrillation: a Mendelian randomization study. Eur J Neurol 2020;27:550-556. https://doi.org/10.1111/ene.14111
- 116. Howe LJ, Tudball M, Davey Smith G, Davies NM. Interpreting Mendelian-randomization estimates of the effects of categorical exposures such as disease status and educational attainment. Int J Epidemiol 2022;51:948-957. https://doi.org/10.1093/ije/dyab208
- 117. Campbell DT. Factors relevant to the validity of experiments in social settings. Psychol Bull 1957;54:297-312. https://doi.org/10.1037/h0040950
- 118. Devesa SS, Blot WJ, Fraumeni JF. Declining lung cancer rates among young men and women in the United States: a cohort analysis. J Natl Cancer Inst 1989;81:1568-1571. https://doi.org/10.1093/jnci/81.20.1568
- 119. Holmes MV, Ala-Korpela M, Smith GD. Mendelian randomization in cardiometabolic disease: challenges in evaluating causality. Nat Rev Cardiol 2017;14:577-590. https://doi.org/10.1038/nrcardio.2017.78
- 120. Richmond RC, Anderson EL, Dashti HS, Jones SE, Lane JM, Strand LB, Brumpton B, Rutter MK, Wood AR, Straif K, Relton CL, Munafo M, Frayling TM, Martin RM, Saxena R, Weedon MN, Lawlor DA, Smith GD. Investigating causal relations between sleep traits and risk of breast cancer in women: Mendelian randomisation study. BMJ 2019;365:l2327. https://doi.org/10.1136/bmj.l2327
- 121. Lundh A, Lexchin J, Mintzes B, Schroll JB, Bero L. Industry sponsorship and research outcome: systematic review with meta-analysis. Intensive Care Med 2018;44:1603-1612. https://doi.org/10.1007/s00134-018-5293-7
- 122. Kesselheim AS, Robertson CT, Myers JA, Rose SL, Gillet V, Ross KM, Glynn RJ, Joffe S, Avorn J. A randomized study of how physicians interpret research funding disclosures. N Engl J Med 2012;367:1119-1127. https://doi.org/10.1056/NEJMsa1202397
- 123. Fabbri A, Lai A, Grundy Q, Bero LA. The influence of industry sponsorship on the research agenda: a scoping review. Am J Public Health 2018;108:e9-e16. https://doi.org/10.2105/AJPH.2018.304677
- 124. Henderson GE, Cadigan RJ, Edwards TP, Conlon I, Nelson AG, Evans JP, Davis AM, Zimmer C, Weiner BJ. Characterizing biobank organizations in the U.S.: results from a national survey. Genome Med 2013;5:3. https://doi.org/10.1186/gm407
- 125. Caulfield T, Burningham S, Joly Y, Master Z, Shabani M, Borry P, Becker A, Burgess M, Calder K, Critchley C, Edwards K, Fullerton SM, Gottweis H, Hyde-Lay R, Illes J, Isasi R, Kato K, Kaye J, Knoppers B, Lynch J, McGuire A, Meslin E, Nicol D, O'Doherty K, Ogbogu U, Otlowski M, Pullman D, Ries N, Scott C, Sears M, Wallace H, Zawati MH. A review of the key issues associated with the commercialization of biobanks. J Law Biosci 2014;1:94-110. https://doi.org/10.1093/jlb/lst004
- 126. Luo S, Au Yeung SL, Zhao JV, Burgess S, Schooling CM. Association of genetically predicted testosterone with thromboembolism, heart failure, and myocardial infarction: Mendelian randomisation study in UK Biobank. BMJ 2019;364:l476. https://doi.org/10.1136/bmj.l476
- 127. Packer M. Data sharing in medical research. BMJ 2018;360:k510. https://doi.org/10.1136/bmj.k510
- 128. Bastian H. “They would say that, wouldn’t they?”: a reader’s guide to author and sponsor biases in clinical research. J R Soc Med 2006;99:611-614. https://doi.org/10.1177/014107680609901207
- 129. International Committee of Medical Journal Editors (ICMJE). Journals stating that they follow the ICMJE recommendations [Internet]. ICMJE; c2021 [cited 2021 Sep 10]. Available from: https://www.icmje.org/recommendations/
- 130. da Costa BR, Cevallos M, Altman DG, Rutjes AW, Egger M. Uses and misuses of the STROBE statement: bibliographic study. BMJ Open 2011;1:e000048. https://doi.org/10.1136/bmjopen-2010-000048
- 131. Caulley L, Catala-Lopez F, Whelan J, Khoury M, Ferraro J, Cheng W, Husereau D, Altman DG, Moher D. Reporting guidelines of health research studies are frequently used inappropriately. J Clin Epidemiol 2020;122:87-94. https://doi.org/10.1016/j.jclinepi.2020.03.006
- 132. Altman DG, Simera I, Hoey J, Moher D, Schulz K. EQUATOR: reporting guidelines for health research. Lancet 2008;371:1149-1150. https://doi.org/10.1016/S0140-6736(08)60505-X
 , Rebecca C. Richmond2,3
, Rebecca C. Richmond2,3 , Benjamin A. R. Woolf2,4
, Benjamin A. R. Woolf2,4 , Neil M. Davies2,3,5
, Neil M. Davies2,3,5 , Sonja A. Swanson6, Tyler J. VanderWeele7
, Sonja A. Swanson6, Tyler J. VanderWeele7 , Nicholas J. Timpson2,3
, Nicholas J. Timpson2,3 , Julian P. T. Higgins3,8
, Julian P. T. Higgins3,8 , Niki Dimou9
, Niki Dimou9 , Claudia Langenberg10,11
, Claudia Langenberg10,11 , Elizabeth W. Loder12,13, Robert M. Golub14,15
, Elizabeth W. Loder12,13, Robert M. Golub14,15 , Matthias Egger1,3,16
, Matthias Egger1,3,16 , George Davey Smith2,3,8
, George Davey Smith2,3,8 , Jeremy Brent Richards17,18,*
, Jeremy Brent Richards17,18,*